Contents
- 1 摘要
- 2 主要
- 3 延长随访的临床结果
- 4 疫苗可诱导长寿命 T 细胞克隆
- 5 长寿命克隆从头启动
- 6 克隆会聚到效应 T 细胞
- 7 疫苗免疫和癌症复发
- 8 讨论
- 9 方法
- 10 数据可用性
- 11 代码可用性
- 12 引用
- 13 确认
- 14 作者信息
- 15 道德宣言
- 16 同行审查
- 17 其他信息:
- 18 扩展数据图表和表格
- 18.1 扩展数据 图 1:在 3 年随访中,Autogene cevumeran 但不相关 atezolizumab 反应或肿瘤大小与 RFS。
- 18.2 扩展数据图 2 Autogene cevumeran 在启动但不增强时诱导从头 T 细胞克隆。
- 18.3 扩展数据 图 3 CloneTrack 鉴定的 Autogene 头孢乌梅兰诱导的克隆包含新抗原特异性克隆。
- 18.4 扩展数据图 4 自基因 cevumeran 诱导的 CloneTrack 克隆的动力学、半衰期和寿命。
- 18.5 扩展数据图 5 共享 TCR Vβ 特异性组中的 T 细胞克隆不与自基因 cevumeran 诱导的 CloneTrack 克隆共扩增。
- 18.6 扩展数据图 6 atezolizumab 诱导的 T 细胞克隆的动力学。
- 18.7 扩展数据图 7 Autogene cevumeran 诱导的 CloneTrack 克隆 T 细胞不会长期获得耗竭特征。
- 18.8 扩展数据 图 8:尽管接种后化疗,Autogene cevumeran 诱导的 CloneTrack 克隆仍保留效应表型。
- 18.9 扩展数据 图 9 自基因头孢乌梅兰诱导的 CloneTrack 克隆频率与体外新抗原特异性功能召回相关。
- 18.10 扩展数据 图 10 自基因头孢乌梅拉反应者不会获得针对非疫苗新抗原的免疫活性。
- 19 补充信息
- 20 源数据
- 21 权利和权限
RNA neoantigen vaccines prime long-lived CD8 T cells in pancreatic cancer+
- 出版日期:
摘要
癌症疫苗的一个基本挑战是产生对肿瘤抗原具有特异性的长寿命功能性 T 细胞。在这里,我们发现针对体细胞突变衍生的新抗原的 mRNA-lipoplex 疫苗可以解决胰腺导管腺癌 (PDAC) 中的这一挑战,PDAC 是一种突变很少的致命癌症。在手术 1 期试验的延长中位随访 3.2 年,atezolizumab(PD-L1 抑制抗体)、自基因头孢莫兰1(具有骨架优化的尿苷 mRNA-脂质体纳米颗粒的个体化新抗原疫苗)和修饰的 (m) FOLFIRINOX(化疗)在 PDAC 患者中,我们发现疫苗诱导的 T 细胞反应者 (n = 8) 的无复发生存期 (RFS;中位数未达到) 与没有疫苗诱导的 T 细胞的无反应者相比 (n = 8;中位 RFS 13.4 个月;P = 0.007)。在应答者中,自基因头孢莫聚糖诱导 CD8 T 细胞克隆,平均估计寿命为 7.7 年(范围 1.5 至大约 100 年),大约 20% 的克隆具有可能比宿主活得更久的潜在数十年寿命。每位患者 86% 的克隆在接种疫苗后约 3 年以相当大的频率持续存在,包括对 PDAC 新表位具有高亲和力的克隆。使用 PhenoTrack(一种追踪单个 T 细胞表型的新型计算策略),我们发现疫苗诱导的克隆在疫苗接种前组织中无法检测到,并在接种疫苗后长达三年内呈现细胞毒性、组织驻留记忆样 T 细胞状态,并保留新抗原特异性效应器功能。两名反应者复发并证明疫苗诱导的 T 细胞较少。此外,从疫苗靶向癌症克隆中修剪了复发性 PDAC。因此,在 PDAC 中,自基因头孢聚糖诱导具有多年寿命、显着幅度和持久效应功能的从头 CD8 T 细胞,可以延迟 PDAC 复发。因此,辅助 mRNA-lipoplex 新抗原疫苗可以解决癌症疫苗接种的关键障碍。++
主要
我们之前报道过1研究者发起的辅助序贯抗 PD-L1(atezolizumab,单次疫苗接种前剂量)的 1 期试验的早期临床和免疫学结果,然后是 8 次静脉内初免剂量的个体化尿苷 mRNA 新抗原疫苗(自基因头孢莫新生;包含多达 20 种主要组织相容性复合体 I 类 (MHCI) 和 II 类 (MHCII) 限制性新抗原,针对主要体细胞乘客突变在 atezolizumab 后 3 周给药), mFOLFIRINOX (12 个周期) 和单次疫苗加强剂量 (扩展数据图 .1a) 在 19 名手术切除的 PDAC 患者中(安全性可评估队列包括 16 名接种疫苗的患者和 3 名接受 atezolizumab 治疗的患者)。在中位随访 1.5 年时,在 16 名接种疫苗的患者(生物标志物可评估队列)中,有 8 名患者自基因头孢梅兰诱导了与延迟 PDAC 复发相关的高强度、多功能新抗原特异性 CD8 效应 T 细胞+1.在这里,我们将临床和免疫学随访时间加倍,以探测疫苗诱导的 CD8 T 细胞是否:(1) 与延迟 PDAC 复发持久相关;(2) 作为推定的记忆 T 细胞长期存在;(3) 长期保留效应器功能。+
延长随访的临床结果
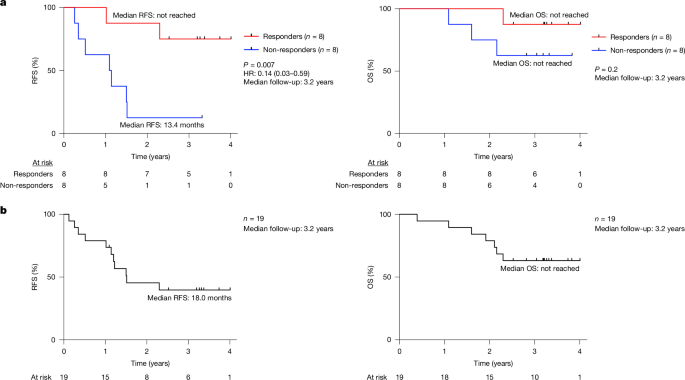
在生物标志物可评估队列中1,中位随访 3.2 年(范围 2.3-4.0 年),通过离体 IFNγ ELISpot 测定的 8 例疫苗诱导的高水平新抗原特异性 T 细胞患者1(响应者)的中位 RFS 未达到(图 D)。1a,左)。相比之下,8 例没有疫苗诱导的 T 细胞患者 (无反应者) 的中位 RFS 为 13.4 个月 (P = 0.007;风险比 = 0.14,95% 置信区间 0.03-0.59;无花果。1a,左)。疫苗反应者和非反应者之间的效应量与 1.5 年中位随访时观察到的效应量相似1,包括通过地标分析来排除响应时间偏倚(Extended Data Fig.1b,左)。与我们之前的发现一致,疫苗反应者不仅富集于对 atezolizumab 有反应的患者或预后较好的患者1,atezolizumab 免疫反应(方法)和肿瘤大小与延迟复发无关(扩展数据图1b,中间和右)。我们之前报道过1反应者和非反应者在整个研究期间具有等效的临床病理预后特征和可比的免疫能力,与同时接种无关 mRNA 疫苗 (SARS-CoV-2) 具有相似的体液和细胞免疫。应答者或无应答者的中位总生存期 (OS) 未达到(图 D)。1a,右)进行长时间随访。在安全性评估队列中,中位 RFS 为 18.0 个月,中位 OS 未达到(图 D)。因此,自基因头孢乌梅拉反应可能与延迟的 PDAC 复发持久相关。
疫苗可诱导长寿命 T 细胞克隆
为了研究疫苗诱导的 T 细胞是否持续存在,我们追踪了所有反应者中疫苗诱导的 T 细胞克隆的来源和寿命(图 D)。为此,我们应用了 CloneTrack1,它利用接种疫苗前后外周血的 T 细胞受体 (TCR) Vβ 测序来检测和追踪高强度疫苗诱导的 T 细胞克隆(方法)。我们之前报道过1在应答者中,CloneTrack 鉴定的克隆几乎都是 CD8 T 细胞,并且包括对疫苗新抗原亚群具有特异性的 CD8 T 细胞,这些细胞在引发后触发阳性离体 IFNγ 酶联免疫斑点反应(8 名应答者的 108 种疫苗新抗原中有 25 种是 ELISpot 阳性++1;无花果。2b,顶部)。
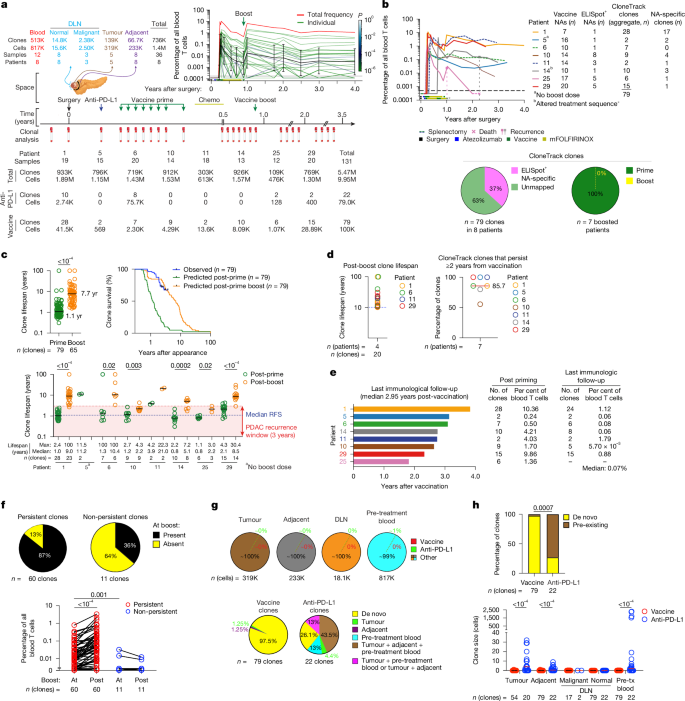
a,分析组织和血液中的 T 细胞克隆。总计包括所有血液 T 细胞和克隆;由 CloneTrack 鉴定的抗 PD-L1 和疫苗克隆。右上,患者 1 的 T 细胞克隆轨迹。抗 PD-L1,阿替利珠单抗;化疗,mFOLFIRINOX;疫苗初免和加强剂量,autogene cevumeran。b,左上,反应者中疫苗诱导克隆的纵向总百分比。彩色矩形表示手术时间、atezolizumab、疫苗接种和 mFOLFIRINOX;星号 (*) 表示患者 14 的治疗顺序改变。水平虚线显示克隆检测的阈值。右上,具有经过验证的新抗原特异性克隆的新抗原 (NA) 数量。下图,映射到 ELISpot 阳性 (ELISpot) 新抗原(左)并由疫苗初免和加强剂量诱导的克隆百分比(右)。c,所有(左上)和个体(下)患者的估计克隆寿命。底部,红色阴影区域表示 3 年历史 PDAC 复发窗口;中位 RFS 是历史术后 PDAC RFS。右上,观察到的克隆存活率与估计的克隆存活率。由于没有 boost 或 clone,没有显示 14 个克隆的 boost 寿命。d,左图,四名具有最长寿克隆的患者的顶部克隆寿命。虚线表示接种疫苗后 10 年。右图为每位患者在接种疫苗后持续两年或更长时间的克隆百分比(持续克隆)。患者 25 在接种疫苗后不到 2 年内死亡。e,最后一次免疫学随访(左;接种疫苗后中位 2.95 年)以及引发后(所有患者)和最后一次随访时(持续性克隆患者)(右)的克隆数量和总百分比。f,克隆(上)和所有血液 T 细胞(下)中 n = 6 名加强患者加强时持续性和非持续性克隆的百分比。g,顶部,含有处理克隆的组织 T 细胞的百分比。底部,组织中包含的处理克隆的百分比。n 是组织 T 细胞(上)和治疗诱导克隆(下)的数量。h,处理前组织(上图)中存在(预先存在)或不存在(从头)的外周血中处理克隆的百分比以及相关的克隆大小(下图)。所有治疗诱导的克隆均通过 CloneTrack 鉴定。前 tx,前处理。n 是克隆、细胞或患者的数量。通过双尾 Mann-Whitney 检验(c、h,底部)、双尾 Wilcoxon 匹配对符号秩检验(f,底部)和双侧卡方检验(h,顶部)得出的 P 值。+
从接种疫苗前到接种疫苗后大约 4 年应用 CloneTrack(26 个时间点的 131 个样品中的 547 万个克隆、995 万个细胞;无花果。2a) 在所有 8 名反应者中,我们确定疫苗在引发后诱导 79 个 T 细胞克隆(“CloneTrack 克隆”;无花果。2b,上图)在所有患者中有多个克隆(扩展数据图 .2a) 的 7 名接受加强剂量的反应者中没有新的加强诱导的克隆(图 D)。2b、底部和扩展数据图通过将同源克隆映射到所有 25 个 ELISpot 阳性疫苗新抗原,我们发现 8 个反应者中有 7 个 79 个 CloneTrack 克隆中有 29 个 (37%) 是 ELISpot 阳性疫苗新抗原特异性 CD8 T 细胞(图 D)。2b、底部和扩展数据图3a-c;其余 50 个 CloneTrack 克隆未定位),包括 5 个反应者中的 15 个克隆,通过 TCR 克隆和最小新表位定位证实了对疫苗新抗原的特异性(扩展数据图 .3a-c)。尽管 CloneTrack 克隆在疫苗剂量数量可变的情况下达到峰值(扩展数据图 .4a) 到不同幅度(扩展数据图4b),77% 的 CloneTrack 克隆在 6 次或更少的引发剂量下达到峰值扩增(扩展数据图 1)。4a),单个克隆扩增了大约 100 倍或更多(扩展数据图 .4c) 的中位峰百分比为所有外周 T 细胞的 0.2%(扩展数据图 .4d,顶部)。因此,在 PDAC 患者中,自基因头孢乌梅兰启动剂量(而不是加强剂量)诱导高幅度的 T 细胞克隆。+
接下来,为了检查尽管患者在引发剂量后立即接受 mFOLFIRINOX 治疗,但 CloneTrack 克隆是否可以长期存在,我们纵向追踪了外周血中克隆随时间的收缩率。对克隆进行深度纵向采样(图 D)。2a),我们可以准确地将标准指数函数拟合到观察到的克隆衰变动力学(扩展数据图 2)。4e,上),从而估计半衰期和寿命(从峰值频率到低于检测阈值的时间;方法)的
疫苗引发后,CloneTrack 克隆的中位寿命估计为 1.1 年(范围 0.3 至约 100 年;无花果。2c,左上),中位半衰期约为 40 天(扩展数据图 D)。4d,底部)。值得注意的是,单次疫苗加强显着将估计克隆寿命延长了 7 倍,中位寿命为 7.7 年,寿命从 1.5 到 100 年不等(或可能超过大约 100 年;方法)(图 .2c,左上角;中位半衰期 297 天;扩展数据 图因此,加强后,7 名患者中有 5 名的中位估计克隆寿命超过了术后 3 年窗口,其中大多数 (约 80%) PDAC 患者历史上复发2,3,4(图 .2c,底部)。值得注意的是,在 7 名加强针患者中,有 4 名克隆(占所有观察到的加强针后克隆的 30%(65 个中的 n = 20 个))估计寿命超过十年(图 D)。2d,左)。峰值膨胀频率与克隆寿命相关(正如指数衰减所预期的那样),但与半衰期无关(扩展数据图 1)。此外,与多个克隆共同扩增的克隆相比,仅用另一个克隆扩增的 CloneTrack 克隆的寿命(初免后)大约长了 7 倍(扩展数据图 .4g),这表明克隆寿命可能会随着竞争克隆的减少而增加。与初免后单位(单一免疫原性疫苗新抗原)反应相比,多位(多重免疫原性疫苗新抗原)患者的克隆寿命略长,但加强后相似(扩展数据图 1)。最后,CloneTrack 克隆寿命反映了新抗原特异性克隆的真实特征,因为 CloneTrack 克隆经实验验证对疫苗中包含的新抗原具有特异性(新抗原特异性;扩展数据 图3a,b)表现出与其他 CloneTrack 克隆相当的半衰期和寿命(扩展数据图 1)。4i)。
为了用观察到的寿命来证实估计的克隆寿命,我们接下来检查了 CloneTrack 克隆是否随着时间的推移而持续存在于外周血中。与观察到的时间范围内的准确指数拟合一致(扩展数据图 1)。4e,下图),与观察到的寿命密切相关的响应者的估计克隆寿命(图 D)。2c,右上角)。此外,在 7 名免疫随访时间超过 2 年的可评估患者中(中位免疫随访时间为接种疫苗后 2.95 年),86%(中位数)的 CloneTrack 克隆(图 D)。2d,右)持续占据所有血液 T 细胞的大部分(中位数 0.07%),包括接种疫苗后约 3.8 年高达 1.1% 的细胞(图 D)。在加强针患者中,接种疫苗后持续 2 年的 CloneTrack 克隆(持续克隆;n = 6 名患者),87% 的患者在加强时存在,而 64% 的非持续性克隆在加强给药前已从循环中消失(图 D)。持久克隆的生成概率更高5 (P根;扩展数据 图5a),估计通过 VDJ 重组生成特定 TCR 的人类库的优先偏斜,表明与非持久性克隆相比,持久性克隆可能包含具有更高生成可能性的 TCR。值得注意的是,疫苗没有共扩增共享 TCR Vβ 特异性组的克隆6使用 CloneTrack 克隆(扩展数据图 .5b,c)。总之,自基因头孢莫聚糖诱导多个 CD8 T 细胞克隆,这些克隆在 PDAC 患者中长期存在,潜在寿命超过 PDAC 复发窗口,并且选择可能比宿主存活更长的克隆。+
长寿命克隆从头启动
为了探寻长寿命 CloneTrack 克隆的来源,我们使用 TCR Vβ 测序在 8 名反应者中的疫苗接种前宿主血液和组织(肿瘤、肿瘤引流淋巴结 (DLN) 和肿瘤相邻的非淋巴组织;736,000 个克隆,36 个样本)中全面搜索 CloneTrack 克隆(图 D)。2a)。
我们只检测到宿主组织中预先存在的 2 个 CloneTrack 克隆中的 2 个细胞:319,000 个肿瘤 T 细胞中有 1 个细胞,来自肿瘤邻近组织的 233,000 个 T 细胞中有 1 个细胞(图 D)。2g,顶部)。在 18,100 个 DLN 或 817,000 个血液 T 细胞中没有疫苗诱导的细胞(图 D)。2g,顶部)。在 79 个 CloneTrack 克隆中,我们只检测到 2.5% 的预处理(图 D)。2g,左下角),总共 736,000 个克隆(140 万个 T 细胞;无花果。相比之下,73% 的抗 PD-L1 扩增克隆(22 个克隆中有 16 个通过 CloneTrack 在疫苗反应者中扩增;扩展数据 图6a) 存在于单个或多个宿主隔室中的预处理,克隆大小可变(图 D)。2g,右下角和图与 CloneTrack 克隆类似,在 58 个新抗原特异性克隆中,只有 6 个在体外鉴定(新抗原特异性加新抗原定位克隆;扩展数据 图3a-c)在 8 名反应者中,任何宿主组织中都已存在(扩展数据图 1。因此,在 PDAC 患者中,与扩增预先存在的克隆的抗 PD-L1 不同,自基因 cevumeran 将疫苗接种前宿主组织中无法检测到的多个幼稚克隆引物到高量级从头。
克隆会聚到效应 T 细胞
由于自基因头孢莫兰诱导多个 T 细胞克隆在血液中长期存在,为了随着时间的推移捕获 CloneTrack 克隆的完整表型历史,我们开发了一种计算策略 (PhenoTrack),该策略利用单细胞 RNA 和 TCR 测序来追踪 9,110 个单 T 细胞的表型,几乎所有(71 个中的 68 个)CloneTrack 克隆在接受疫苗初免和加强剂量的反应者中,其中克隆持续到大约 3 个接种疫苗后几年。
首先,我们注意到,如前所述,在患者中,CloneTrack 克隆是1,主要是 CD8 T 细胞(扩展数据图 1)。7a),其转录特征根据疫苗接种后克隆扩增(扩增阶段,疫苗启动期间)、收缩(收缩阶段,疫苗启动结束至 mFOLFIRINOX 启动)的时间分离,并且预计会作为记忆 T 细胞持续存在+7(记忆期、收缩后;无花果。3a,左)。相对于所有其他阶段,扩增期的 CloneTrack 克隆过表达增殖细胞的基因特征(MKI67 和其他基因集富集分析(方法);增殖状态),而效应基因(GZMB、GZMH、NKG7、PRF1 和 JUN 家族基因;效应态)在收缩期受到调节(图 D)。3a,右和补充表 1)。相比之下,持续在记忆期的 CloneTrack 克隆不会差异性地过表达中枢或效应记忆 T 细胞的常规标志物8(包括 SELL、TCF1、IL7R、CCR7 等;扩展数据 图7b) 而是过表达效应基因 (GZMB、GZMM、GZMH、GNLY、NKG7 和 JUN 家族基因) 和 CD8 组织驻留记忆 T 的核心转录因子 (T+马币)细胞 (ZNF683 (也称为 HOBIT);T马币-like 状态;无花果。3a,右侧和补充表 1)9—驻留在组织中并在抗原再攻击时表现出强效效应功能的长寿命 T 细胞9,但也可能循环10并抑制处于低抗原负荷状态的休眠癌细胞11.这些发现表明,自基因头孢乌梅兰引发的 T 细胞早期增殖,然后收缩,保留效应特征并在可能的 T 中长期存在马币-样状态,具有循环能力。
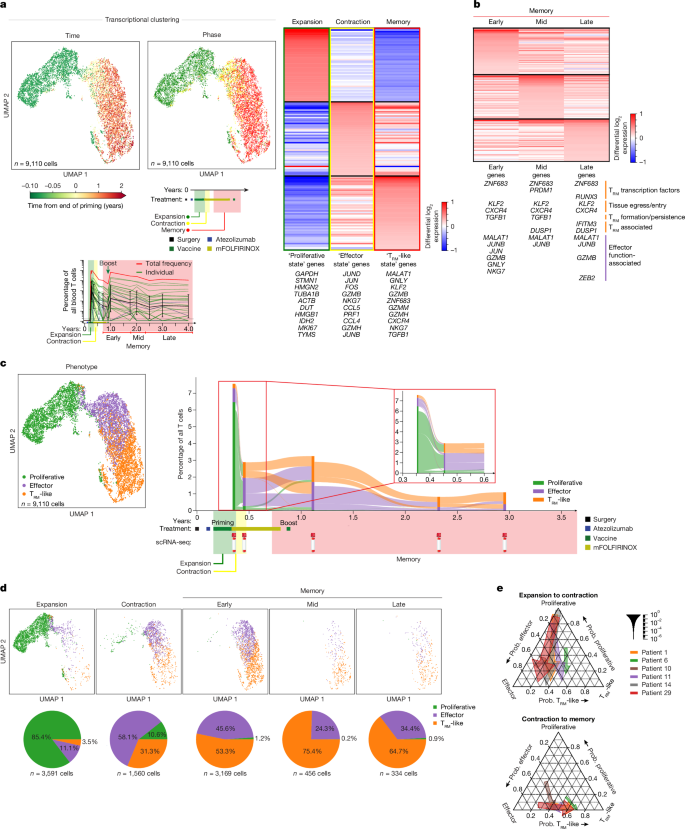
a–e,通过单细胞 RNA 和 TCR 测序,来自 6 名疫苗反应者血液的 71 个 CloneTrack 克隆中有 68 个的纵向表型。a,左上,按疫苗接种后时间和阶段划分的 CloneTrack 克隆 T 细胞的均匀流形近似和投影 (UMAP) 图。左下角是患者 1 的 CloneTrack 克隆阶段示意图。a,b,按相对于其他相的相位(a,右)和相对于其他子相的记忆子相(b),CloneTrack 克隆 T 细胞中排名前 50 位的差异表达基因。基因列表包括具有增殖、效应和 T 特征的基因马币按期或亚期过表达的细胞(补充表 1 中提供了完整的基因列表)。c,左图,单细胞 RNA 测序 (scRNA-seq) 数据中按表型对 CloneTrack 克隆 T 细胞进行转录聚类。右图为 6 名响应者中 71 个 CloneTrack 克隆中 68 个克隆随时间变化的表型组成和转化率的 PhenoTrack 图。垂直彩色条形显示相应时间每种表型的细胞比例。单个 PhenoTrack 图显示在 Extended Data Fig 中。8b. d,疫苗接种后 CloneTrack 克隆 T 细胞的 UMAP(上图)和表型组成(下图)。e,在 n = 6 个具有持久克隆的引发和增强反应者中,CloneTrack 克隆从扩增到收缩(上)和收缩到内存(下)的表型转换。表型绘制为每个响应者克隆的每种表型的概率 (prob.)。患者 25 没有持续克隆,并在图 1 中进行了单独分析。5a. 患者 5 未接受加强。箭头宽度表示跨患者汇总的克隆频率;箭头基部与轴的接近程度表示表型中每个患者的细胞分数;箭头从表型起点指向目的地。n 是单元或克隆的数量。
由于记忆期跨越了接种疫苗后的 2.5 年,我们接下来将记忆期细分为早期(0.8-1.8 年)、中期(1.9-2.3 年)和晚期(超过 2.4 年)记忆期(图 D)。3b 和补充表 1)进一步探究 CloneTrack 克隆是否获得了 T马币类似功能。与 T 对齐马币具有循环能力的样表型,相对于所有其他阶段,CloneTrack 克隆不仅过表达核心 T马币转录因子基因 ZNF68312在所有记忆阶段中选择性地表达,但也逐渐共过表达其他两个标志 T马币转录因子基因 PRDM112(也称为 BLIMP1)和 RUNX313在后面的记忆阶段(图此外,记忆期的 CloneTrack 克隆也过表达转录因子基因 KLF2,该基因促进组织出口8,14、促进组织进入的 CXCR4、可能促进 T 的 TGFB1马币形成和持久性8,15 元和 T 细胞干性8(图 .3b)、低表达的组织保留受体(扩展数据图 .7c) 和逐渐保留和过表达的效应子 (GZMB、JUN 家族、ZEB2 和 MALAT1)16、17、18 元等 T马币相关(IFITM3 和 DUSP1)19,20 元基因 (图 .3a,b)。记忆期的 CloneTrack 克隆在 T 细胞耗竭连续体上不表达表型标记物8,21 元(扩展数据图 .总的来说,这些数据强烈表明自基因头孢梅聚糖诱导 CD8 T 细胞在循环中长期存在,并且在稳定的非耗竭 T 中持续存在+马币类似效应子的状态。
接下来,由于 CloneTrack 克隆表现出三种不同的疫苗接种后状态(扩增期间的增殖状态、收缩期间的效应子状态和 T马币-like 状态;无花果。3a),我们试图检查单个克隆表型如何随时间转变。我们将单细胞分配给与增殖、效应和 T 相关的表型马币-like 状态(图3c,左,扩展数据图8a 和方法)和使用 Sankey 图可视化克隆表型转换(图 .图 3c,右图和扩展数据图8b),它与标准 Sankey 和冲积图的不同之处在于跟踪克隆在膨胀和收缩时的表型。
在扩增阶段,85% 的 CloneTrack 克隆细胞处于增殖状态,11% 处于效应状态,3.5% 处于 T 状态马币-like 状态(图 .然后克隆在 5 周内迅速收缩并过渡(图 D)。3c,右插图)从增殖状态到主要效应子(58% 的疫苗诱导的 T 细胞)和 T马币-样(31% 的疫苗诱导的 T 细胞)处于收缩阶段的状态(图 D)。3d),其中单个 CD8 T 细胞克隆的细胞具有所有表型(图 D)。3e、顶部和扩展数据图8c,顶部)。进入记忆期,克隆保持两种细胞状态——尽管它们主要是 T+马币在接下来的 2.5 年里,尽管患者在初免和随后的加强剂量后接受了 mFOLFIRINOX,但仍保留了效应器特征(图 D)。3c,d)。与增殖到收缩的转变一样,单个克隆同时在两种细胞状态下持续存在,从收缩到记忆(图 D)。3e、底部和扩展数据图8c,底部)。总之,自基因头孢聚糖诱导 CD8 T 细胞克隆从头长期收敛到 T+马币样和效应 T 细胞,它们可能准备对抗原再攻击做出反应。
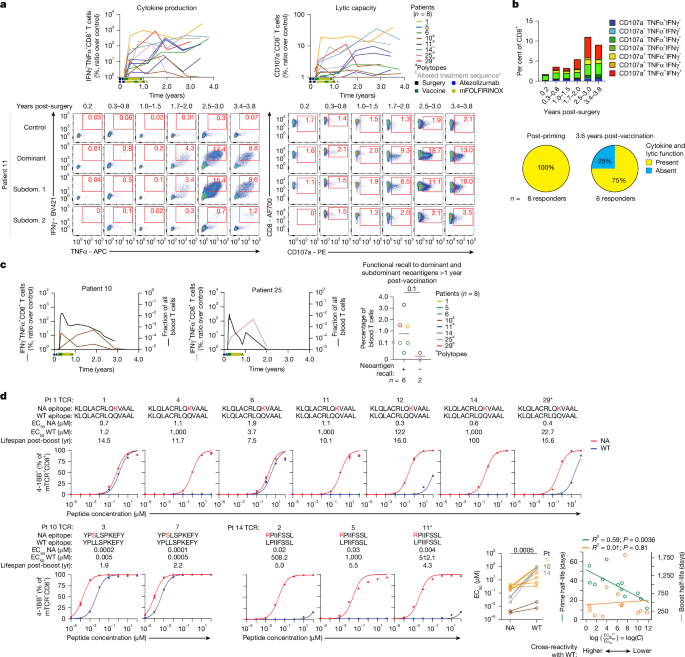
为了测试 CloneTrack 克隆在反应者血液中的持久性是否与长期保留的新抗原特异性功能回忆相关,我们在体外连续重新攻击在引发后至接种疫苗后约 3.6 年(手术后约 3.8 年)分离的外周血单核细胞 (PBMC),并检查 CD8 T 细胞细胞因子的产生和裂解能力(图总体而言,所有疫苗反应者中的 CD8 T 细胞产生细胞因子(IFNγ 和 TNFα)并在引发后新抗原再攻击时脱颗粒(图 D)。4a) 并在 8 名疫苗反应者中的 6 名接种疫苗后长达 3.6 年内继续产生效应细胞因子并显示出裂解潜力(图 D)。4a,b)。具体来说,在 4 个单位应答者中有 4 个(扩展数据图 .9a) 和 4 个多位体应答者中的 2 个(图 .4a、bottom、c 和扩展数据图9b),CD8 T 细胞继续产生 IFNγ 和 TNFα,并在体外新抗原再攻击后脱颗粒,先前测定+++1免疫斑点阳性显性和次显性疫苗新抗原在接种疫苗后长达 3.6 年。此外,新抗原功能回忆的丧失与克隆持久性大致相关,因为两名没有回忆的患者在接种疫苗后后期表现出血液中 CloneTrack 克隆总频率最低的多位反应(患者 10 和 25;无花果。4c,右),这表明持续存在的 CloneTrack 克隆可以介导效应器功能。为了进一步表征长寿命疫苗诱导克隆的特征,我们绘制了 12 个 TCR 的亲和力,包括 10 个 CloneTrack 克隆和 2 个在体外鉴定的疫苗新抗原特异性克隆(扩展数据图 1)。3b) 在 3 名反应者中长期持续(中位寿命 8.8 年)(图疫苗诱导克隆的 TCR 显示较低的半数最大有效浓度 (EC50) 和对同源新表位的亲和力高于野生型表位,并且包括具有纳米和亚纳摩尔亲和力的新表位-TCR 对(图 .4d,右下角),证实 PDAC(一种突变负荷低的肿瘤)中的乘客突变可以产生可被内源性 T 细胞识别的高亲和力新表位。此外,与野生型表位相比,TCR 对新表位的相对亲和力更高(更高的对数 (C))与引发后较短的克隆半衰期相关,但与增强后不相关(图 D)。4d),表明较高的相对表位强度可能与克隆腐烂的速度相关。总之,这些结果表明,在 PDAC 患者中,自基因头孢莫兰诱导 CD8 T 细胞克隆,包括对 PDAC 新表位具有高亲和力的克隆,这些克隆在接种疫苗后持续长达 3.6 年,长期保留多种效应功能,并显示出持续数十年的潜力。+
a-c,免疫显性和亚显性 (subdom) 体外再攻击后 CD8 T 细胞的纵向细胞因子产生和裂解能力。反应者中的 ELISpot 阳性疫苗新抗原。a,反应者中 IFNγTNFαCD8 T 细胞(细胞因子产生)和 CD107aCD8 T 细胞(裂解能力)的上图再攻击后总百分比。y 轴显示新肽活化的 T 细胞组分与 DMSO 激活的 T 细胞组分的比率。星号 (*) 表示患者 14 的治疗顺序改变。下图是来自疫苗反应者的 T 细胞的代表性流式细胞术。b,体外新抗原再攻击后 CD8 T 细胞亚群的纵向中位百分比(上)和多功能(细胞因子CD107a)疫苗新抗原特异性 CD8 T 细胞反应者百分比(下)。c,左图,两名免疫力减弱的反应者在体外再攻击后血液中 CloneTrack 克隆 T 细胞的总分数和 IFNγTNFαCD8 T 细胞的百分比。右图:接种疫苗后一年多,通过体外再攻击,按有或没有新抗原特异性 IFNγTNFαCD8 T 细胞的反应者分层的血液中 CloneTrack 克隆 T 细胞的总百分比。a-c,显性和次显性新抗原是先前确定的疫苗新抗原++++++++++++++++1在疫苗启动后 1-3 周收集的 PBMC 中通过离体 IFNγ 酶联免疫斑点诱发 IFNγ T 细胞(显性新抗原引发最大 T 细胞反应;次显性抗原引发非最大 T 细胞反应)。以 DMSO 为对照。d、Avidity(左)和 EC+50与人白细胞抗原 (HLA) 匹配、新肽或野生型 (WT) 肽脉冲抗原呈递细胞一起培养的 TCR 转导的 CD8 T 细胞的值(右)。从 CloneTrack 或新抗原特异性克隆(星号)中分离的 TCR 标识如 Extended Data Fig.3a-c.红色显示的残基是突变的氨基酸。log(C) 是抗原距离+28.流式细胞术在活体 CD3CD56 上设门+−CD8 细胞 (a) 或活的 CD3CD8 TCR 转导的细胞 (d)。n 是患者数。通过双尾 Mann-Whitney 检验(c,右)和双侧 Wilcoxon 匹配对符号秩检验(d,右下)得出的 P 值。d 中的误差线表示 s.e.m.+++
疫苗免疫和癌症复发
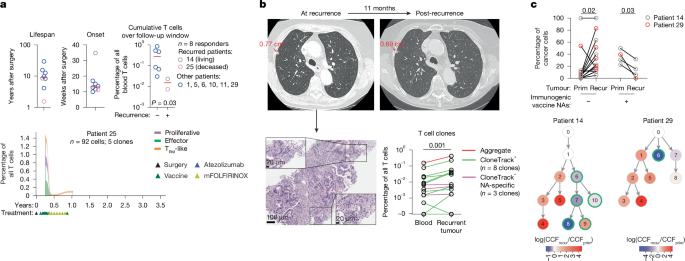
由于两名反应者 (患者 25 和 14) 在观察期间复发,我们探讨了疫苗免疫力的变化是否与 PDAC 复发相关。患者 25 — 多位反应者、第一个复发反应者和唯一死亡反应者(来自局部复发性 PDAC)— 与其他反应者相比,观察到的 T 细胞寿命短了大约 10 倍(图 D)。5a,左)。所有 6 个 CloneTrack 克隆在 8 次引发剂量后急剧下降(扩展数据图 .值得注意的是,在该患者中,加强剂量重新扩大,但没有挽救引发的 CloneTrack 克隆,随后 CloneTrack 克隆在患者死亡前消失(扩展数据图 D)。这种较短的 T 细胞寿命并不是由于不同的后启动表型(图 2)。5a,底部)。与其他响应者类似(图3c,e 和扩展数据图8b,c),来自该患者的 CloneTrack 克隆是引发后增殖和效应表型的混合物,收敛到 T马币-like 状态。患者 14 — 单位反应者和第二反应者复发(稳定、孤立的 1 cm 肺转移)— 治疗顺序改变,导致 CloneTrack 克隆起病晚(术后 34 周)与其他 7 名反应者(中位术后 13 周;无花果。5a,中间),克隆同样采用效应子和 T马币-like 状态(扩展数据图 .因此,复发的两个个体的累积疫苗免疫力较弱(在随访窗口整合的聚合 CloneTrack 克隆 T 细胞;无花果。5a,右),因为寿命最短或发病最晚。
a,上图,反应者在观察期内观察到的疫苗诱导的 CloneTrack 克隆 T 细胞的估计寿命(左)、观察时间(中)和累积频率(右),无论是否复发。下图,患者 25 中 CloneTrack 克隆 T 细胞的纵向表型。b,患者 14 孤立性肺转移的计算机断层扫描(上图;肿瘤直径为红色)和组织病理学(左下)。右下,血液中疫苗诱导的 T 细胞克隆和患者 14 的复发性肿瘤。CloneTrack,由 CloneTrack 识别的血液 T 细胞克隆,如 Extended Data 图 所示。2a;克隆跟踪+−NA 特异性血液 T 细胞克隆在体外被鉴定为新抗原特异性,如扩展数据图 所示。3a-c.c,携带免疫原性和非免疫原性疫苗新抗原的原发性和复发性肿瘤中癌症克隆的百分比(上图)和系统发育(下图)(通过离体 IFNγ ELISpot 确定1) 在患者 14 和 29 中。原发性原发性肿瘤;复发性、复发性肿瘤。红色和蓝色分别表示从原发性肿瘤到复发性肿瘤的癌症克隆分数 (CCF) 的增加和减少。绿色圆圈表示具有免疫原性疫苗新抗原的克隆。n 是患者、细胞或克隆的数量。双尾 Mann-Whitney 检验(a,右)、双尾 Wilcoxon 匹配对符号秩检验(b,下)和双尾配对 t 检验(c,上)的 P 值。
为了检查 CloneTrack 克隆是否保留了浸润复发性肿瘤的功能,我们在患者 14 的孤立性肺转移中搜索了 CloneTrack 克隆(图 1)。5b,顶部)。与血液相比,在记忆晚期持续存在的 8 个 CloneTrack 克隆中,有 7 个在复发性肿瘤中克隆扩增(图 D)。5b,底部)。此外,三个新抗原特异性克隆在体外鉴定,但未通过 CloneTrack 鉴定(扩展数据图 .3b) 也存在于复发性肿瘤中(图 D)。5b),表明 CloneTrack 和体外鉴定的新抗原特异性克隆都保留了体内浸润远处肿瘤的功能。为了进一步检查这些 T 细胞克隆是否对临床上检测不到的肿瘤克隆施加选择压力,我们测试了疫苗诱导的免疫是否可以对携带免疫原性疫苗新抗原的肿瘤克隆施加选择压力,无论是直接通过疫苗靶向肿瘤克隆的选择性丢失,还是间接通过对未包含在疫苗中的新抗原的获得性免疫。我们没有检测到针对肿瘤突变新抗原的获得性免疫活性(这表明新表位扩散)的明确证据,这些新抗原未包含在任何反应者的疫苗中(扩展数据图 .然而,使用全外显子组测序和系统发育分析,我们检查了原发性 PDAC 如何纵向演变为患者 14 和患者 29(先前报道的患者)的复发1发展为由疫苗诱导的 T 细胞克隆浸润的消失孤立性肝微转移(未满足临床复发标准)。与疫苗诱导的免疫对微转移性肿瘤克隆施加选择性压力一致,两名患者的复发性肿瘤进化为携带免疫原性疫苗新抗原的肿瘤克隆较少(图 D)。5c)。
讨论
我们能否克服宿主耐受性,产生针对肿瘤抗原的强壮、特异性、功能性和耐用的 T 细胞?如果是这样,哪些抗原、疫苗和宿主可以共同产生这样的反应?在 PDAC 中,这是最致命和最具肿瘤学挑战性的癌症之一,它导致 88% 的患者死亡22,我们表明 autogene cevumeran 是一种基于尿苷的未修饰的 mRNA-lipoplex 疫苗,针对体细胞突变衍生的新抗原,在疾病负担最小的患者中给药,可能为这一挑战提供复合解决方案。
CloneTrack 在固定初免-加强疫苗接种后鉴定的疫苗诱导的 CD8 T 细胞克隆的长寿和功能值得注意。疫苗诱导的 CloneTrack 克隆具有估计的多年寿命,近四分之一的克隆估计具有数十年的寿命,比保守估计的患者寿命长。值得注意的是,这些细胞在接种疫苗后长达 3.6 年内表现出强大的新抗原特异性 T 细胞多功能性。此外,疫苗诱导的克隆可以浸润复发的 PDAC,正如之前在一名患者中观察到的那样+1在这项研究的一秒钟内。尽管一名患者(患者 1)为疫苗诱导的克隆库贡献了大量克隆,但对个体患者的亚群分析揭示了所有个体加强患者中克隆的大量估计寿命。这些寿命估计值将在未来的工作中需要额外的实验验证。然而,它们表明,在患病宿主中无需重复增强即可产生对肿瘤抗原具有特异性的长寿命、功能性 CD8 T 细胞。我们的疫苗接种策略和 mRNA 疫苗平台似乎超过了几种疫苗试金石——疫苗诱导的 CD8 T 细胞在稳态下持续存在的大小似乎与在高度成功的病原体疫苗中观察到的疫苗诱导的 CD8 T 细胞的大小相当,甚至更高+++23,即使是单次免疫也能赋予终生免疫力24.此外,疫苗诱导的长期免疫主要由 CD8 T 细胞组成,模拟了 PDAC 中可能的“自然”免疫控制+25.然而,我们不能排除疫苗也会诱导 CD4 T 细胞。至关重要的是,疫苗诱导的免疫跨越了 PDAC 复发窗口,因此有可能在整个最高易感期内提供保护,这是癌症疫苗的理想特征。尽管我们证明靶向最小病变宿主中新抗原的未修饰 mRNA 脂质复合物可触发持久免疫,但针对其他抗原类别或利用新兴疫苗平台的疫苗是否能够实现类似的持久功能还有待观察。+
我们不能排除疫苗诱导的 T 细胞克隆可能存在于本研究中未采样的肿瘤或淋巴结区域。然而,值得注意的是,尽管 PDAC 的突变负荷相对较低,因此其假定的新抗原含量较低,但自基因头孢莫兰(一种基于尿苷的 mRNA 新抗原疫苗)从头引发长寿命 T 细胞对抗乘客突变,而不是扩增预先存在的 CD8 T 细胞。鉴于此类新抗原是肿瘤发生的基本副产品,并且对病情轻微的宿主进行辅助治疗是一种普遍存在的癌症治疗范式,我们的研究结果将理论上符合疫苗条件的池扩展到几乎所有癌症,而不仅仅是那些引起强烈内源性新抗原特异性 T 细胞反应的癌症。免疫学上突变较少的冷肿瘤(例如胃肠道癌症)似乎是理想的初始疾病适应症,因为突变负荷通常相对较低,对免疫检查点抑制剂不敏感,持续依赖上一代细胞毒性化疗作为标准治疗以及预后不良。+
尽管我们发现较低的累积疫苗免疫力与反应者的早期复发相关,但这一观察的意义受到样本量小的限制。尽管如此,该观察结果进一步支持了辅助自基因 cevumeran 可能延迟 PDAC 复发的假设。事实上,我们在 3.2 年随访中报告了疫苗反应与 RFS 之间的持久相关性,其效应量与早期 1.5 年随访中观察到的效应大小相似1.虽然我们之前没有检测到1(目前选择性地确认)反应者和非反应者治疗、肿瘤或宿主免疫适应性、未知因素的差异可能会混淆疫苗反应与 RFS 之间的相关性。尽管如此,利用 6 名患者中 9,000 多个单细胞的完整克隆历史,我们表明大多数疫苗诱导的 CD8 T 细胞克隆作为稳定的效应 T 细胞转变为记忆期,并提供了 mRNA 癌症疫苗诱导的 T 细胞的纵向状态转变和长期表型的全面表征。值得注意的是,尽管多次启动剂量,但持续到记忆期的疫苗诱导的 T 细胞不会获得耗竭特征+26,并且可能在表型上类似于人类 T马币小鼠中描述的具有循环功能的细胞10.值得注意的是,疫苗诱导的新抗原特异性功能的丧失与纵向 T 细胞表型变化无关,但似乎与新抗原特异性 T 细胞本身的丧失有关。尽管尚不清楚疫苗诱导的 T 细胞是否在肿瘤内保留了这种效应功能,但这一观察结果为通过实时监测和预定或按需增强来维持或挽救免疫力提供了可能性。尽管是推测性的,但用于动态监测和增强 T 细胞的疫苗 T 细胞“滴度”——正如我们所证明的,使用 CloneTrack 是可行的——可以推动这种进步,并值得未来的研究。
在疫苗诱导的选择压力下进化的复发性 PDAC 的突变谱既可预测又值得注意。虽然我们没有观察到这种克隆修剪以及 T 细胞免疫扩散到未接种疫苗的突变新抗原,但这一发现表明疫苗诱导的 T 细胞靶向亚临床癌症克隆,因此,疫苗耐药的一种机制可能是克隆逃逸。值得注意的是,缺乏观察到的新抗原传播并不排除免疫扩散到其他突变或非突变抗原,因为我们研究中测试的新抗原可能是非免疫原性的。事实上,在新抗原负荷较高的情况下,新抗原播散可能更明显27,例如在引起更强内源性免疫的更高突变癌症中,并且在突变较少的肿瘤中可能不是一个观察到的特征。尽管如此,有效的癌症疫苗可能需要针对所有异质性癌症克隆的初始多价疫苗,随后进行新抗原重采样和适应性疫苗接种以靶向克隆逃逸,或者使用诱导 T 细胞对抗克隆新抗原的疫苗,具有高效力和持久性。
总之,我们通过更长时间的随访提供了额外的证据,证明自基因头孢乌梅拉(一种针对体细胞突变衍生的新抗原的基于尿苷的 mRNA 疫苗)可以在 PDAC 中产生与延迟复发相关的强、特异性、功能性和持久的 CD8 T 细胞免疫。一项全球随机试验 (IMCODE 003;NCT05968326)正在进行中。由于突变衍生的新抗原是转化细胞的基本特征,我们的研究结果为 mRNA 新抗原疫苗作为辅助癌症疗法的广泛测试提供了支持。+
方法
试验设计、患者和终点
在我们之前报道的1单中心、研究者发起的 1 期临床试验,根据我们的计算管道预测的单个、放射学可疑、可手术切除的 PDAC、无远处转移和 ≥5 新抗原的患者接受序贯手术、辅助阿替利珠单抗(抗 PD-L1)、自基因头孢莫兰(一种基于尿苷 mRNA-脂质复合物纳米颗粒的个体化疫苗,编码高达 20 MHCI 和 MHCII 限制性新抗原)和 mFOLFIRINOX。所有患者的 ECOG (Eastern Cooperative Oncology Group) 体能状态为 0-1。我们纳入了经病理证实的 PDAC 且手术切缘状态为 R0/R1 的患者。我们排除了转移性、临界性或局部不可切除的 PDAC 患者以及接受新辅助治疗的患者。主要终点是安全性。次要终点是 18 个月的 RFS 和 18 个月的 OS。其他资格标准、程序、治疗和伦理研究行为已经描述,并在方案中提供1(补充文件 1)。
我们用 atezolizumab 治疗了 19 例患者 (安全性可评估队列),其中 16 例患者接受了后续的自体基因 cevumeran (生物标志物可评估队列)1.这 16 例患者中有 15 例随后接受了 mFOLFIRINOX。如前所述1,如果接种疫苗的患者对疫苗新抗原产生高幅度的 T 细胞反应,则被归类为自基因头孢乌梅兰(疫苗)反应者,该反应由先前描述的离体 IFNγ ELISpot 测定评估29检测疫苗诱导的 T 细胞反应,并且不区分 CD8 和 CD4 T 细胞反应。++
我们根据实体瘤的反应评估标准(RECIST,1.1 版)将复发定义为新病灶。我们定义了从手术日期 (RFS) 或最后一次自基因头孢梅拉启动剂量 (里程碑式 RFS) 到复发或死亡日期的 RFS,以先发生者为准。我们定义了从手术日期到死亡日期的 OS。我们审查了在最后已知日期无复发且无事件的患者。我们将复发窗口定义为 ~80% 的患者复发的手术时间范围2,3,4.数据截止日期为 2023 年 12 月 1 日,中位随访时间延长至 38 个月。
我们根据赫尔辛基宣言和良好临床实践指南进行了这项研究。该研究已获得美国联邦药物管理局 (FDA) 纪念斯隆凯特琳癌症中心 (MSK) 机构审查委员会 (IRB) 的批准,并于 clinicaltrials.gov (NCT04161755) 注册。所有参与者都提供了书面知情同意书。
个性化疫苗的突变鉴定和新抗原选择
我们之前报道了疫苗新抗原选择的详细特征1.简而言之,我们通过患者特异性肿瘤-正常对的全外显子组测序和肿瘤 RNA 测序鉴定了表达的非同义突变和 HLA 类型。然后,我们用生物信息学估计了新抗原,并如前所述按免疫原性对新抗原进行了排名30.
Autogene cevumeran 生产
据报道1,对于每位患者,我们在良好生产规范条件下生产了个体化 mRNA 新抗原疫苗,作为两条基于尿苷的 mRNA 链,具有非编码序列,经过优化以实现卓越的翻译性能31,32 元.我们设计了每条链来编码多达 10 种 MHCI 和 MHCII 新抗原,这些新抗原配制在直径约为 400 nm 的脂质体纳米颗粒中26它包括合成阳离子脂质 (R)-N,N,N-三甲基-2,3-二烯氧基-1-丙胺氯化铵 (DOTMA) 和磷脂 1,2-二油酰-sn-甘油-3-磷脂酰乙醇胺 (DOPE),以实现静脉内输送。
患者样本、细胞系和细胞培养物
我们通过在 Ficoll-Paque Plus (GE Healthcare) 上进行密度离心纯化患者 PBMC,并在补充有 10% 胎牛血清(FBS、Nucleus Biologics)、1 mM 丙酮酸钠、2 mM l-谷氨酰胺、非必需氨基酸和 2-巯基乙醇的 RPMI 培养基中培养纯化细胞,100 U ml−1青霉素 – 链霉素(MSK 培养基制备核心设施),含 100 U ml−1IL – 2 (Peprotech) 和 100 U ml−1每隔一天添加一次 IL-15 (Peprotech)。为了绘制单克隆反应性图,我们购买了泛阴性选择纯化的人 T 细胞(Precision for Medicine)和在补充有 10% FBS (Sigma-Aldrich)、100 U ml 的 RPMI 培养基(Gibco,Thermo Fischer Scientific)中培养的纯化 T 细胞−1青霉素 – 链霉素(Gibco,Thermo Fischer Scientific),1,000 U ml−1IL-7(Peprotech,Thermo Fischer Scientific),100 U ml−1IL-15(Peprotech,Thermo Fischer Scientific)和 4 mM l-谷氨酰胺(Gibco,Thermo Fischer Scientific)。我们用补充有 10% FBS(Sigma-Aldrich)和 100 U ml 的 RPMI 培养基(Gibco,Thermo Fischer Scientific)培养 T2 和 K562 细胞−1青霉素 – 链霉素(Gibco,Thermo Fischer Scientific)。我们用补充有 10% FBS (Sigma-Aldrich) 和 100 U ml 的 DMEM 培养基(Gibco,Thermo Fischer Scientific)培养 Phoenix-AMPHO 和 RD114 包膜生产细胞−1青霉素 – 链霉素(Gibco,Thermo Fischer Scientific)。我们从切除的肿瘤标本中冷冻肿瘤、邻近组织和引流淋巴组织样本以进行后续分析。
HLA 克隆和转导
如前所述,我们克隆了患者匹配的 HLA 等位基因并转导了 T2 和 K562 细胞1.简而言之,我们将 HLA 等位基因和 IRES GFP 报告基因克隆到 SFG γ-逆转录病毒载体中,并使用 Lipofectamine 3000 (Thermo Fisher Scientific) 转染 Phoenix-AMPHO 细胞。我们收集了含有载体的上清液,并在 Polybrene (EMD Millipore) 存在下通过离心接种使用它们转导 T2 和 K562 细胞。我们使用 Aria 细胞分选仪 (BD Biosciences) 对 HLA K562 细胞和 GFP T2 细胞进行分选。++
TCR 重建、克隆、转导和肽刺激
如前所述,我们重建和克隆了 TCR1.简而言之,我们从纯化、测序的单个 T 细胞中鉴定了 T 受体 β (TRB) V-D-J 和 T 受体 α (TRA) V-J 序列,并将它们分别与修饰的小鼠恒定 TRB 和 TRA 链序列融合。然后,我们将 TRB 和 TRA 链与弗林蛋白酶 SGSG P2A 接头连接起来,并将它们克隆到 SFG γ-逆转录病毒载体中。我们如前所述转导了人 pan T 细胞1稍作修改。简而言之,我们使用 Lipofectamine 3000 (Thermo Fisher Scientific) 用 SFG γ-逆转录病毒载体转染 Phoenix-AMPHO 细胞。然后,我们使用含有载体的上清液转导 RD114 包膜生产细胞。我们使用 Retro-X 浓缩仪 (Takara) 对转导的细胞进行分类,以生成稳定的细胞系和浓缩的含载体的上清液。我们用 CD3/CD28 珠子 (Thermo Fisher) 激活人 pan T 细胞,并在激活后第 3 天使用浓缩的含载体的上清液在 Retronectin (Takara) 包被板中转导它们。通过对活 CD8 mTCR 细胞进行流式细胞术门控来确认转导效率。++
为了选择可能的新抗原表位来测试 TCR 特异性和结合性,我们从所有全长 (18-27 聚体) 免疫原性(ELISpot 阳性)疫苗新抗原中生成了所有可能的独特 8-14 聚体。我们预测了这 8 到 14 聚体中的每一个都通过 NetMHCPan4.1 与患者的 HLA-I 结合。我们进一步选择了结合亲和力为 <1000 nM 或预测的强结合剂(根据 NetMHCPan4.1)的肽,用于进一步测试和肽刺激。
我们用肽刺激 TCR 转导的 T 细胞,如下所示:我们脉冲 2.5 × 105HLA 转导的 T2 或 K562 细胞在 37 °C 的 96 孔 U 底板中,用指定浓度的指定肽保存 1 小时。然后,我们离心并洗涤肽,加入 5 × 104TCR (效应子:靶标比为 1:5) 或模拟 (对照) 每孔转导的 T 细胞,并在共培养 24 小时后测量 CD8 小鼠 TCR T 细胞上的 CD137 (4-1BB) 表达。为了计算 TCR 对其同源表位和相应野生型肽的亲和力,我们用用不同浓度的突变或野生型肽脉冲的抗原呈递细胞刺激 TCR 转导的 T 细胞(范围从 100 μM 到 10++−5μM),执行非线性回归以将数据拟合为 S 形曲线,并计算 EC50作为激活 50% TCR 转导的 T 细胞的肽浓度。当 TCR 未显示与 EC 的反应性时50值趋于无限,我们分配了 EC50为 1,000 μM。
免疫应答
TCR Vβ 测序
我们使用 DNA 提取试剂盒 (Qiagen) 从大量 PBMC、纯化的 T 细胞和患者组织样本中提取 gDNA。我们使用 Dropsense 96 定量样品、稀释标准品浓度并制备文库。然后,我们使用 immunoSEQ 检测 (Adaptive Biotechnologies) 生成样本数据,如前所述1.简而言之,我们使用两步扩增偏倚控制的多重 PCR 反应扩增体细胞重排的 TCRB CDR3,该反应包含对每个已知 V 和 J 基因片段具有特异性的正向和反向扩增引物,并扩增 CDR3 受体高变位点,然后进行第二次 PCR 以添加条形码和 Illumina 接头序列33.PCR 中的参考基因引物进一步定量可用于测序的总有核细胞,从而测量每个测序样品中 T 细胞的分数。然后对 CDR3 和参考基因文库进行测序,根据 Adaptive 的条形码序列对原始序列读数进行多路分离,并对分离读数进行处理以去除接头和引物序列,并识别和去除引物二聚体、种系和污染物序列。使用相似克隆之间的相对频率比和改进的最近邻算法对过滤结果进行聚类,并合并密切相关的序列以纠正测序引起的技术错误。然后将输出序列注释到构成每个独特 CDR3 的 V、D 和 J 基因以及 N1 和 N2 区域,以及相应的编码 CDR3 氨基酸序列。我们根据 IMGT 数据库 (https://www.imgt.org) 定义了注释基因。然后对输出的 TCR V CDR3 序列进行归一化和校正残留多重 PCR 扩增偏倚,并与合成的 TCRB CDR3 序列类似物进行定量34.
单细胞 RNA 和 TCR 测序
据报道1,我们在 Weill Cornell Medicine 的表观基因组学核心准备了用于单细胞免疫分析、测序和后处理原始数据的文库。
样品制备
为了分析疫苗诱导克隆的基因表达,通过荧光激活细胞分选纯化来自患者来源的 PBMC 的大量 T 细胞,并按照先前报道的方式进行测序1.简而言之,在进行单细胞免疫分析之前,我们根据公司规范纯化了单 T 细胞并制备了 scRNA-seq 文库(10x Genomics,指南 CG000330)。将每个活力为 97% 且每 μl 有 950 个细胞的细胞悬液上样到铬 X 上,以生成靶向每个样品 10,000 个单细胞的凝胶微珠乳液 (GEM)。
测序和数据处理
为了生成 5 P 表达文库,我们将 cDNA 的等分试样 (~50 ng) 酶促片段化,并进行末端修复,A 尾,使用 SPRI 选择珠 (Beckman Coulter) 进行双面大小选择,并连接到接头。然后,我们通过 14 个 PCR 扩增循环(98 °C 持续 45 秒;98 °C 持续 20 秒,54 °C 持续 30 秒和 72 °C 持续 20 秒× 14 个循环;72 °C 持续 1 分钟;保持在 4 °C),对索引进行第二次双面大小选择, 以及使用 Qubit 荧光定量 (Thermo Fisher Scientific) 的定量文库。我们在 Agilent Bioanalyzer 2100 上评估了质量,获得的平均文库大小为 460 bp。为了生成全长 TCR VDJ 区域,我们将 cDNA 等分试样 (5 ng) 与特异性 VDJ 外部和内部引物对(98 °C 45 秒;98 °C 20 秒,67 °C 30 秒和 72 °C 20 秒× 8 个循环;72 °C 1 分钟;保持在 4 °C),进行巢式 PCR 扩增, 使用 SPRI select 微珠进行单侧大小选择,并使用 Agilent Bioanalyzer 2100 评估 VDJ 区域的质量和数量。平均文库大小为 620 bp。
然后,我们将基因表达和 TCR 文库聚集在对 28-10-10-91 测序的 Illumina Novaseq 对末端流动槽上,每个样品获得约 3.5 亿个簇。我们使用 Illumina 的实时分析软件 (RTA) 处理测序图像。我们使用 10x Genomics Cell Ranger 单细胞软件套件 v6.0.0 (https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/latest/what-is-cell-ranger) 对样本进行多路分离、对齐 (hg19)、过滤、UMI 计数和单细胞 5′ 末端基因计数样本,组装 TCR,注释配对的 VDJ,并根据制造商的参数进行质量控制。
克隆跟踪
克隆定义
我们通过 β 链的核苷酸 CDR3 序列及其 V 和 J 基因 (TRB) 定义了 T 细胞克隆。对于通过单细胞测序鉴定的 T 细胞,我们通过 CDR3B 核苷酸序列定义了克隆,并将克隆映射到配对的 TCR Vβ 测序。忽略 V/J 基因以消除 V/J 基因调用中的跨平台偏倚。
我们将克隆 x 的频率定义为 \({f}_{x}\,=\frac{{n}_{x}}{N}\),其中 nx是对应于上述克隆 X 的读取数,N 是所有生产性(框内,无终止密码子)读取的总和,如前所述1.
我们使用聚合计数 \({n}_{X}={\sum }_{x\in X}{n}_{x}\) 计算了几个克隆 \(x\in X\) 的总频率,并使用了前面描述的伪频率1如果 \({n}_{X}=0\) 。
如前所述,我们定义了疫苗诱导和 atezolizumab(抗 PD-L1)诱导的克隆1.简而言之,在 CloneTrack 中,我们选择一个基线时间点和一个感兴趣的时间段,并计算每个克隆的 P 值,以确定它在确定的时间段内是否扩展了两倍或更多,使用前面描述的修改后的 Fisher 精确检验1,对克隆和检查的时间点数进行 Bonferroni 多重假设检验调整。我们为每个克隆选择最显著的时间点,调整后的 P < 0.001。对于疫苗诱导的 CloneTrack 克隆,我们使用疫苗启动剂量之前的时间点作为基线,并定义延长至化疗第一剂(完成所有启动剂量后)的扩展时间段。我们对引物克隆施加了从头约束,因此在引物剂量开始之前不得在血液中检测到克隆。对于抗 PD-L1 扩增的克隆,我们使用抗 PD-L1 剂量之前的时间点作为基线,并定义扩增时间段以延长至抗 PD-L1 之后的时期。我们更新的分析产生的克隆与之前检测到的克隆相同1.
我们通过修改上述修改后的 Fisher 精确检验定义了由疫苗加强诱导的克隆1在考虑 Bonferroni 多重假设校正后,提升前后的时间点之间具有两倍扩展因子,以及从头约束和调整后的 P < 0.001 的显著性阈值。未发现诱导疫苗加强克隆。
半衰期和寿命
由于我们在 CloneTrack 时间序列中有足够的时间点,我们可以利用标准数学方法来估计生物和物理系统中的半衰期和寿命——将指数函数拟合到实验观察到的单个 T 细胞克隆的衰变率。对于每个克隆,我们绘制了实验测量的轨迹(扩展数据图 .4e,上图),然后得出单独引发剂量和引发 + 加强剂量后克隆的半衰期和寿命,如下所述。
- (1)为了估计克隆的半衰期和启动后的寿命,我们确定了峰值启动时间(克隆的频率在增强之前达到峰值的时间),并选择从峰值启动到增强的所有时间(扩展数据图 D)。4e,顶部,橙色圆圈)。
- (2)为了估计克隆的半衰期和初注/增强后的寿命,我们确定了峰值增强时间(克隆在增强后首次测量频率的时间),并选择峰值增强后的所有时间。
当一个克隆没有被检测到时(频率 = 0),我们估算它的频率 \({f}_{{\rm{imputed}}}=0.5/\max \_{\rm{sample}}\_{\rm{norm}}\),其中 \(\max \_{\rm{sample}}\_{\rm{norm}}\) 是时间序列中最大测序样本的总序列数。
然后,我们假设克隆的频率随时间呈指数衰减:
其中 f(t) = 时间 t 的克隆频率;f0= 克隆频率峰值;τ = 平均寿命。有两个自由参数 \(f(t)\) 和 \({t}_{1/2}\),必须推断出来。为了推断每条曲线的自由参数,我们使用最小二乘回归(使用 stats.linregress 实现)对克隆频率的对数进行了线性拟合。
我们通过从所有后续时间中减去峰值时间来得出 \({t}_{0}=0\),并将指数方程线性化:
我们现在可以将线性回归拟合到上述方程中,以得出衰减常数 \(\tau \)。拟合斜率为 \(m=-\,\frac{1}{\tau }\)。因此:
由于克隆的半衰期是克隆从其峰值频率降低 50% 的时间,
其中 m 是观察到的轨迹的拟合斜率。
我们将生命周期定义为克隆频率低于检测阈值 (10−6).我们推导出寿命 (t消失) 设置为:
我们从这些拟合中报告的两个量是半衰期 (\({t}_{1/2}\)) 和寿命 (t消失).然后将这些估计值裁剪为半衰期 \({t}_{1/2}\) 的最大值为 5 年,消失时间 \(\Delta {t}_{d}\) 的最大值为 100 年,包括任何正在生长而不是缩小的克隆,这将推断出负半衰期和寿命。
为了估计拟合优度,我们计算了测量每个克隆的均方根误差 (RMSE),即每个估计点在该时间点与观察到的频率的距离,并在时间序列中平均。
具体来说,RMSE 的计算公式为
其中 N 是要拟合的点数,\({y}_{i}^{{\prime} }\) 是时间点 i 和 y 的估计频率我是在时间点 i 观察到的频率。
我们在拟合方程的自然对数空间中计算 RMSE,并将 RMSE 可视化(扩展数据图 .4e,底部)在原木上10scale,相当于我们绘制克隆轨迹的比例尺(图 D)。2b 和扩展数据图所有估计频率都在观测频率的 10 倍以内,144 个估计频率拟合中的 98 个 (68%) 位于观测值的 2 倍以内。
CloneTrack 生存曲线
CloneTrack 克隆的存活曲线是根据克隆首次观察的时间确定的。由于所有这些克隆在疫苗接种前的血液中均未观察到,因此此出现时间 (t一个) 可以精确确定。对于观察到的存活时间 (\({t}_{s}\)),我们取了从最后一次观察到克隆的时间点到出现时间的差值。对于估计的生存曲线,我们使用消失时间 \(\Delta {t}_{d}\) 和开始时间 \({t}_{0}\) (峰值起始频率或增强后随访时间)来确定该拟合的生存时间:
请注意,患者 5 没有接受加强,因此他们的两个 CloneTrack 克隆都包含在主要和总估计生存曲线中。
时间积分累积疫苗接种频率
为了测量 CloneTrack 克隆在设定时间点的累积频率,我们计算了聚合 CloneTrack 频率时间序列的积分平均值。由于 T 细胞克隆频率在对数刻度上变化,我们对频率轨迹的对数进行积分和平均,然后在最后取指数(加权几何平均值);
其中 \({p}_{c}(t)\) 是 CloneTrack 克隆 c 的频率轨迹。为避免采用 0 的对数,我们使用了 2 × 10 的最小频率−6如果在某个时间点未测量 CloneTrack 克隆。我们使用时间 t = 644 天进行分析,对应于所有患者都可以进行完整克隆分析的最新随访时间点。使用 scipy.integrate.cumtrapz 完成数值积分。
元克隆
为了找到可能对疫苗有反应的其他 T 细胞克隆,我们寻找了具有与疫苗反应性克隆(即 CloneTrack 克隆)相似的 TCR 序列的克隆型。我们使用了 GLIPH2.035使用默认参数对每位患者的所有 β 链 TCR(包括 CDR3 氨基酸序列、V 和 J 基因)进行聚类,使用该患者最可用的 TCR 测序样本(将样本限制为早期随访)。与 GLIPH2 提供的背景 CD4/CD8 参考 TCR 相比,在标记为显著的 GLIPH 组 (Fisher 评分 <0.05) 中,我们确定了那些包含 CloneTrack 克隆 (疫苗反应元克隆) 的组。对于每位患者,在每个时间点鉴定出具有 CDR3 氨基酸序列的 TCR 克隆(由其核苷酸序列和 V/J 基因定义),并绘制了这些克隆(不包括原始 CloneTrack 克隆)的频率总和所有可用时间点。
单细胞分析
质量管理
使用 Scanpy 将 Cellranger (v6) 生成的过滤计数矩阵集成到单个矩阵中36.通过从 Cellranger VDJ (v6) 调用测序的高置信度 TRB CDR3 区域来过滤细胞。根据基因表达在表达基因的细胞集(非零表达)上的分布是否至少具有 1 nat 的熵,并且该基因是否在至少 1% 的总细胞中被发现,进一步过滤基因。线粒体和核糖体基因均被去除。
表型
使用 GeneVector 流程进行表型37.通过质量控制的细胞和基因根据 GeneVector 方法使用相关系数签名的互信息嵌入 20 维潜在空间38.然后对属于 CloneTrack 克隆的细胞进行表型分析(由其 TRB 序列识别),方法是将具有三个分量的高斯混合模型与每个细胞包埋向量的余弦相似性拟合到嵌入六个向量(MKI67、GZMB、ZNF683 和 KLF2)的五个基因上,如 Scikit-Learn 中实现的那样39.
PhenoTrack
我们使用上述表型方法生成了 PhenoTrack 图。每个 PhenoTrack 图都是通过检查特定个体的一组 C 个 T 细胞克隆来定义的,并跟踪每个克隆的相对频率和表型流。由于克隆可以在库中扩展或消失,因此这些图与标准的 Sankey 和冲积图的不同之处在于,节点的流入和流出没有平衡。为了定义这些图,我们首先计算了每个表型 f 的频率我对于每个克隆 c 在每个时间点 T, \({p}_{c,T}(\,{f}_{i}\,))\)。这可以表示为具有克隆型 c 的所有单元格 x 的总和(表示为 \(x \sim c\)):
其中 XT是时间点 T、N 的测序克隆集T是 X 中的单元格总数T,并且 \(p(\,{f}_{i}\,|x)\) 是单元格 x 具有表型 f 的概率我(如果使用确定性赋值,则为 0 或 1)。每个测序时间点的静态表型细分可以通过对 C 语言中的所有克隆求和来轻松确定:
这由序列点处的实心条表示。我们永远无法确定单个细胞的表型交换,因为任何经过测序的细胞都必然不存在于下一个时间点,因此为了确定从一个点到下一个点的表型流动,我们检查了克隆的表型概率。同样重要的是要注意,由于克隆 c 在时间点 T 的频率可能与在时间点 T + t 的频率不同,因此从一个时间点流出的总流量可能不等于流入下一个时间点的流量。这就需要我们定义相邻时间点之间的流出和流入。表型从表型 f 中流出我到 fj对于时间点 T 和 T + t 之间的单个克隆 c 可以根据独立近似值定义:
此相同过渡的流入仅在克隆频率规范化方面有所不同:
要获得克隆集 C 上的总流数,我们只需要对克隆 \(c\in C\) 求和:
和
这些流出和流入,以及两者之间的简单 Hill 函数插值,决定了显示表型之间转换的条带的宽度。每个条带都根据它流出的表型进行着色。另请注意,由于克隆可能不在两个时间点上,因此一个时间点的总流入/流出可能不等于静态概率,即:
和
表示克隆在这些相应的时间点离开或进入库。对于在个体上聚集的 PhenoTrack 图,所有这些方程都是在个体上平均的。
我们对疫苗接种前/疫苗接种后阶段的定义如下:
- 扩展:自基因 cevumeran 启动剂量期间的所有时间段
- 收缩:自基因头孢梅拉引发剂量后和 mFOLFIRINOX 之前的所有时间段
- 早期记忆:接种疫苗后 0.8-1.8 年
- 中期记忆期:接种疫苗后 1.9-2.3 年
- 晚期记忆期:2.4 年以上的所有时间段
三元图
通过将表型空间减少到三个表型并仅就这些表型确定每个感兴趣克隆的表型细分来生成三元图:
然后,这些 3D 概率向量可以绘制在 2D 单纯形上。单个时间点可以表示为散点图,时间点之间的转换可以用箭头表示。标记的宽度或大小表示克隆在初始时间点的频率。还可以通过求和和重新规范化将克隆聚合在一起。
P 根
每个氨基酸 CDR3 TRB 通过 VDJ 重组生成的概率,也称为 P根,是使用 OLGA 软件计算的40.P根使用默认人类 TRB IGoR 模型为响应患者的所有基线样本中的每个 TRB 计算41随 OLGA 一起发货。每个 P根计算氨基酸 CDR3 TRB,不包括 V 和 J 基因限制。
基因表达分析
对于差异基因分析,我们只专门检查了患者 1、6、10、11、14 和 29 (n = 9,110 个细胞) 中属于 CloneTrack 克隆的细胞。我们通过每个细胞中的计数总数(由 scanpy.normalize_total 实现)对基因表达进行标准化,并计算标准化基因表达的对数基因表达(由 scanpy.logp1 实现):
其中 GE(c,g) 是细胞 c 中基因 g 的读取计数。
我们逐相计算了阶段间的差异基因热图。仅考虑非核糖体或线粒体的基因 (n = 24,800)。对于每个阶段,我们使用 Mann-Whitney U 和 Bonferroni 多重假设校正,调整后的 P 值阈值为 0.001,确定了相对于所有其他 (CloneTrack) 细胞在该阶段的细胞中上调最显着的基因。然后,通过显著性的基因按其平均差异对数进行排序2基因表达:
其中 Ph 表示来自特定阶段的细胞。我们将扩增、收缩、记忆等阶段的前 25 个上调基因描绘为热图,该热图由上述每个阶段每个基因的差异表达组成(图 D)。3d)。
我们通过使用基因 g 的 LGE(c,g) 为标记相的细胞生成了该基因 g 的基因表达小提琴图。
我们进行基因集富集分析,以确认疫苗诱导的 T 细胞在扩增阶段表达的基因受到增殖 T 细胞的调节42(基因组:Travaglini_Lung_Proliferating_NK_T_Cell,P 值 = 2.3 × 10−22,错误发现率 q 值 = 3.7 × 10−19),以及抗原暴露后由 CD8 T 细胞调节的收缩期 (基因集:Kaech_Day8_Eff_VS_Memory_CD8_TCell_DN,P 值 = 7.8 × 10+−18,错误发现率 q 值 = 5.4 × 10−15).
全外显子组测序、突变鉴定和系统发育
我们应用了与前面描述的相同的生物信息学管道28.简而言之,我们使用 BWA (v0.7.17) 将读数与参考人类基因组 (hg19) 进行比对43,并通过 picard-2.11.0 MarkDuplicates (https://broadinstitute.github.io/picard/) 标记重复项。然后,我们通过基因组分析工具包 (GenomeAnalysisTK-3.8-1) 进行了插入缺失重对齐44使用 RealignerTargetCreator 和 IndelRealigner。我们使用 1000 个基因组 phase1 插入缺失 (1000G_phase1.indels.b37.vcf) 和 Mills 插入缺失检出 (Mills_and_1000G_gold_standard.indels.b37.vcf) 作为参考。然后,我们使用 dbSNP 138 版作为参考源,通过 GATK 的 BaseRecalibrator 校准碱基质量。经过预处理步骤后,我们使用了 MuTect 1.1.745和 Strelka 1.0.1546与正常组织相比,鉴定肿瘤样本中的 SNV 和 indels。使用 bam-readcounts (v0.8) (https://github.com/genome/bam-readcount),我们计算了每个突变位置的参考和替代等位基因的数量。然后,我们根据以下标准按覆盖率和变异等位基因频率 (VAF) 过滤突变:(1) 肿瘤总覆盖率 ≥10;(2) 肿瘤 VAF ≥4%;(3) 肿瘤具有替代等位基因 ≥9 的读数数;(4) 普通≥7 的总覆盖率;(5) 正常 ≤1% 的 VAF。过滤后,我们利用原始样本和复发样本,对每个个体体内过滤的突变进行联合。这使我们能够捕获从原发性肿瘤到复发性肿瘤的覆盖率和 VAF 变化,甚至是未检测到的突变。我们对这组联合突变重复了 bam-readcount 步骤。由于 VAF 不是表示癌细胞分数 (CCF) 的标准化指标,我们使用了 phyloWGS (v1.0-rc2)47重建系统发育树,并在合并的突变集上推断 CCF。原发性和复发性肿瘤具有相同的拓扑结构,但原发性和复发性样本之间的 CCF 可能不同。我们为每个样本选择了具有最大似然值的树。我们使用 R 包 data.tree (v1.1.0) (https://cran.r-project.org/web/packages/data.tree/index.html) 可视化了树木。颜色表示 CCF 从原发性肿瘤到复发性肿瘤的对数刻度变化(对数 CCF娱乐/CCF拘谨).
体外肽刺激
对于每种新抗原,我们设计了 2-4 个重叠的 15 聚体肽 (OLP,Genscript),覆盖了由 RNA 疫苗编码的突变新抗原序列 (Genscript)。我们将所有肽重悬于 DMSO 中,浓度为 10 mg ml−1.然后,我们培养 1 × 106PBMC 在具有肽池的 48 孔板中(总肽浓度为 20 μg ml)−1)和IL-2(100 U ml−1)和 IL-15 (10 ng ml−1) 在第 2 天和随后的每 2-3 天添加一次。然后,在第 7 天,我们用肽库重新刺激 PBMC,并通过用抗 CD107a 抗体孵育受刺激的细胞 1 小时来分析 T 细胞脱颗粒,并通过在37°C 下用蛋白质转运抑制剂 (BD Biosciences) 孵育受刺激的细胞 4 小时来分析 T 细胞 TNFα 和 IFNγ 的产生,并通过流式细胞术测量细胞内细胞因子的产生。
表位扩散
为了检测反应者中的表位扩散,我们在体外用疫苗中未编码的新抗原刺激 PBMC。首先,我们使用 WES 来识别如上所述的体细胞突变。然后,我们过滤掉疫苗中包含的突变,以低置信度调用,或者其转录本编码患者参考蛋白质组中存在的氨基酸序列(即,我们排除了同义突变、停止增益突变和起始损失突变)。从这组中,我们计算创建了 27 聚体,突变残基位于第 14 位(在突变残基之前或之后可用的氨基酸少于 13 个,包括所有可用的残基),测试了 27 聚体衍生的 8 到 14 聚体新表位(不包括所有未跨越突变位点的 k-mer)与患者 HLA-I 的结合通过 NetMHCPan4.1, 并选择了 27 个聚体,其中包含至少一个预测为强结合剂(根据 NetMHCPan4.1)或具有结合亲和力的新表位27<150 纳米。然后,我们设计了 2-4 个 OLP (Genscript),覆盖突变的 27 聚体,并如上所述对大量 PBMC 进行体外肽刺激。
流式细胞术和细胞分选
我们使用 FACSDiva 软件 (版本 8.0.1,BD Bioscience) 在 FACS LSR Fortessa (BD Biosciences) 上分析 PBMC 和转导的 T 细胞。我们根据制造商的建议使用抗体混合物对细胞表面和细胞内标志物进行细胞表面和细胞内标志物染色,最终染色体积为 100 μl,并按指示使用适当的对照。我们使用了以下抗体和试剂:来自 BioLegend 的 CD137(克隆 4B4-1,PE;每个样品 3 μl)、CD3(克隆 SK7、PE-Cy7;每个样品 4 μl)、CD4(克隆 OKT4,亮紫 650;每个样品 2 μl)、CD45(克隆 2D1,Alexa Fluor 700;每个样品 5 μl)、CD56(克隆 HCD56、BV605;每个样品 2 μl)、CD8(克隆 SK1、 AF700 和 APC-Cy7;每个样品 2 μl)、HLA_ABC(克隆 W6/32,APC;每个样品 3 μl)、IFNγ(克隆 4S。B3,BV421;每个样品 5 μl)和 mTCR(克隆 H57-597,PE-Cy5;每个样品 0.5 μl);来自BD Biosciences的CD107a(克隆H4A3,PE;每个样品20 μl)、CD56(克隆NCAM16.2,BV786;每个样品5 μl)、TNFα(克隆Mab11,APC;每个样品2.5 μl)和DAPI(564907;每个样品0.2 μl);以及来自 Invitrogen 的可固定活性染料 eFluor 520(65-0867-14;每个样品 1.0 μl)。我们使用以下定义:人类 T 细胞为活的 T 细胞,CD45CD56+−CD3 细胞;将 CD8 T 细胞脱颗粒为活细胞,CD56++−CD3CD8CD107a;新肽刺激的 CD8 T 细胞为活细胞,CD56++++−CD3CD8TNFαIFNγ;和活化转导的 T 细胞为活的 CD3CD8mTCR4-1BB。我们使用 Aria 细胞分选仪 (BD Biosciences) 对 T 细胞进行分选。我们使用 FlowJo(第 10 版)分析了数据。++++++++
通过体外新抗原再攻击鉴定 T 细胞克隆
如前所述鉴定体外新抗原特异性 T 细胞克隆1稍作修改。简而言之,为了确定 T 细胞克隆是否受到新肽池的特异性刺激,我们将纯化的 T 细胞克隆分为活化和灭活组分(CD107a 和 CD107a+−) 进行 TCR Vβ 或单细胞 VDJ 测序的肽刺激后。
为了鉴定抗原特异性 T 细胞克隆,我们使用具有阈值 S 的单尾二项式测试(实施 scipy.stats.binom_test)确定了每个 T 细胞克隆的肽特异性刺激 P 值,以指示相对于克隆的至少 S 分数的显着性被抗原激活。我们设置阈值,以便通过以 0.1 的步长扫描从 0 到 1 的所有阈值,在实验的对照 (DMSO) PBMC 中未检测到扩增的 T 细胞克隆。选择在每个 DMSO 对照中不扩增的最小阈值,然后添加额外的 0.1 步长,以增强实验中鉴定的扩增克隆的更高特异性。我们根据 TRB 连接的核苷酸序列定义了克隆。我们使用 Bonferroni 校正调整了 P 值,并在调整后的 P 值阈值 <0.001 下确定了显着性。
统计分析
样本量 (n) 表示患者、样本、克隆或细胞的数量。我们通过对数秩 (Mantel-Cox) 检验分析生存曲线,使用未配对的双尾 Mann-Whitney 检验比较两组,使用双尾配对 t 检验或双尾 Wilcoxon 匹配对符号秩检验的配对组,使用 Kruskal-Wallis 检验的多组,通过卡方检验分析分类变量,以及与 Spearman 相关性的相关参数。我们通过双尾 Fisher 精确检验比较了纵向克隆扩增,并通过二项式试验与 Bonferroni 校正比较了体外克隆激活。对于差异基因表达分析,我们使用 Wilcoxon 秩和,没有任何多重假设校正。P < 0.05 被认为具有统计学意义。所有分析均使用 GraphPad Prism (版本 10.1.1) 或 Python (版本 3.11.6) 进行。
报告摘要
有关研究设计的更多信息,请参阅本文链接的 Nature Portfolio Reporting Summary。
代码可用性
CloneTrack 的代码可在 https://github.com/zsethna/CloneTrack 获取。PhenoTrack 的代码可在 https://github.com/zsethna/PhenoTrack 获取。识别 GLIPH2 元克隆的代码可在 http://50.255.35.37:8080/tools 获得。
引用
Rojas, LA 等人。个性化 RNA 新抗原疫苗刺激胰腺癌中的 T 细胞。自然 618, 144–150 (2023)。
Neoptolemos, JP 等人。胰腺癌切除术后氟尿嘧啶加亚叶酸与吉西他滨的辅助化疗:一项随机对照试验。美国医学会 304, 1073–1081 (2010)。
Uesaka, K. 等人。S-1 与吉西他滨辅助化疗治疗切除的胰腺癌:一项 3 期、开放标签、随机、非劣效性试验 (JASPAC 01)。柳叶刀 388, 248–257 (2016)。
Neoptolemos, JP 等人。吉西他滨和卡培他滨辅助治疗吉西他滨单药治疗胰腺癌切除患者的比较 (ESPAC-4):一项多中心、开放标签、随机、3 期试验。柳叶刀 389, 1011–1024 (2017)。
Murugan, A., Mora, T., Walczak, A. M. & Callan, C. G. 从序列库中统计推断T细胞受体产生概率。美国国家科学院院刊 109, 16161–16166 (2012)。
Glanville, J. 等人。识别 T 细胞受体库中的特异性组。自然 286, 958 (2017)。
Sallusto, F., Lanzavecchia, A., Araki, K. & Ahmed, R.从疫苗到记忆,再回到。免疫力 33, 451–463 (2010)。
Gebhardt, T., Park, S. L. & Parish, I. A. 癌症中的干细胞样耗竭和记忆CD8 T细胞。癌症 23, 780–798 (2023)。+
Buggert, M., Price, D. A., Mackay, L. K. & Betts, M. R. 人类循环和组织驻留记忆CD8 T细胞。Nat. 免疫学。 24, 1076–1086 (2023 年)。+
Fonseca, R. 等人。发育可塑性允许常驻记忆 T 细胞产生由外而内的免疫反应。Nat. 免疫学。 21, 412–421 (2020 年)。
Park, SL 等人。组织驻留记忆 CD8 T 细胞促进皮肤中的黑色素瘤免疫平衡。自然 565, 366–371 (2019)。+
Mackay, L. K. 等人。Hobit 和 Blimp1 指示淋巴细胞中组织驻留的通用转录程序。科学 352, 459–463 (2016)。
Milner, J. J. 等人。Runx3 对 CD8 T 细胞在非淋巴组织和肿瘤中的驻留进行编程。自然 552, 253–257 (2017)。+
Carlson, CM 等人。Kruppel 样因子 2 调节胸腺细胞和 T 细胞迁移。自然 442, 299–302 (2006)。
Crowl, J. T. 等人。组织驻留记忆 CD8 T 细胞对不同的组织环境具有独特的转录、表观遗传和功能适应。Nat. 免疫学。 23, 1121–1131 (2022 年)。+
Kragten, N. A. M. 等人。Blimp-1 诱导 CD8 T 细胞中细胞毒性介质颗粒酶 B 并维持 Hobit 维持。Eur. J. 免疫学杂志。 48, 1644–1662 (2018)。
NR4A 转录因子限制实体瘤中的 CAR T 细胞功能。自然 567, 530–534 (2019)。
Wheeler, BD 等人。lncRNA Malat1 抑制 miR-15/16 以增强细胞毒性 T 细胞活化和记忆细胞形成。eLife 12, RP87900 (2023)。
Wakim, L. M., Gupta, N., Mintern, J. D. & Villadangos, J. A. 由于IFITM3的选择性表达,在流感病毒感染期间增强了肺组织驻留记忆CD8 T细胞的存活率。Nat. 免疫学。 14, 238–245 (2013 年)。+
Ostkamp, P. 等人。单细胞分析框架允许表征多发性硬化症中的 CSF 白细胞及其起源组织。Sci. Transl. Med. 14,EADC9778 (2022)。
Giles, J. R., Globig, A.-M., Kaech, S. M. & Wherry, E. J. 癌症免疫周期中的CD8 T细胞。免疫力 56, 2231–2253 (2023)。+
Siegel, R. L., Miller, K. D., Wagle, N. S. & Jemal, A. 癌症统计,2023年。Ca. Cancer J. Clin. 73, 17–48 (2023 年)。
Akondy, R. S. 等人。接种疫苗后人类记忆 CD8 T 细胞的起源和分化。自然 552, 362–367 (2017)。
Marraco, S. A. F. 等人。黄热病疫苗接种后具有幼稚样特征的长效干细胞样记忆 CD8 T 细胞。Sci. Transl. Med. 7, 282RA48 (2015 年)。+
Balachandran, VP 等人。鉴定胰腺癌长期幸存者的独特新抗原特性。自然 551, 512–516 (2017)。
Kranz, LM 等人。向树突状细胞的全身 RNA 递送利用了癌症免疫治疗的抗病毒防御。自然 534, 396–401 (2016)。
胡, Z. 等人个人新抗原疫苗在黑色素瘤患者中诱导持续记忆 T 细胞反应和表位扩散。Nat. Med. 27, 515–525 (2021 年)。
Łuksza, M. 等人。新抗原质量可预测胰腺癌幸存者的免疫编辑。自然 606, 389–395 (2022)。
BNT162b2 疫苗在人体中诱导中和抗体和多特异性 T 细胞。自然 595, 572–577 (2021)。
Sahin, U. 等人。个性化 RNA 诱变组疫苗动员针对癌症的多特异性治疗免疫。自然 547, 222–226 (2017)。
Holtkamp, S. 等人。抗原编码 RNA 的修饰可提高树突状细胞的稳定性、翻译功效和 T 细胞刺激能力。血液 108, 4009–4017 (2006)。
Kreiter, S. 等人。突变的 MHC II 类表位驱动对癌症的治疗性免疫反应。自然 520, 692–696 (2015)。
Robins, H. 等人。对稀有 T 细胞克隆进行超灵敏检测。J 免疫方法 375, 14–19 (2012)。
Carlson, CS 等人。使用合成模板设计无偏倚的多重 PCR 检测。Nat. Commun. 4, 2680 (2013 年)。
Huang, H., Wang, C., Rubelt, F., Scriba, TJ & Davis, M. M. 通过G细胞受体聚集与GLIPH2和全基因组抗原筛选来分析结核分枝杆菌免疫反应。Nat. 生物技术。 38, 1194–1202 (2020)。
Wolf, F. A., Angerer, P. & Theis, F. J. SCANPY:大规模单细胞基因表达数据分析。基因组生物学。 19, 15 (2018 年)。
Ceglia, N. et al. TCRi:癌症中单细胞 RNA 和 TCR 测序的信息论指标。bioRxiv https://doi.org/10.1101/2022.10.01.510457 (2022) 的预印本。
Ceglia, N. 等人。使用 GeneVector 的互信息定义的密集载体表示来识别转录程序。Nat. Commun. 14, 4400 (2023 年)。
Pedregosa, F. 等人。Scikit-learn:Python 中的机器学习。arXiv https://doi.org/10.48550/arxiv.1201.0490 (2012) 的预印本。
Sethna, Z., Elhanati, Y., Callan, CG, Walczak, A. M. & Mora, T. OLGA: 快速计算B细胞和T细胞受体氨基酸序列和基序的产生概率。生物信息学 35, btz035- (2019)。
Marcou, Q., Mora, T. & Walczak, A. M. 使用 IGoR 进行高通量免疫库分析。Nat. Commun. 9, 561 (2018 年)。
Travaglini, KJ 等人。来自单细胞 RNA 测序的人肺分子细胞图谱。自然 587, 619–625 (2020)。
Li, H. 用 BWA-MEM 比对序列读取、克隆序列和组装重叠群。arXiv https://doi.org/10.48550/arxiv.1303.3997 (2013) 的预印本。
McKenna, A. 等人。基因组分析工具包:用于分析下一代 DNA 测序数据的 MapReduce 框架。基因组研究 20, 1297–1303 (2010)。
Cibulskis, K. 等人。灵敏检测不纯和异质性癌症样本中的体细胞点突变。Nat. 生物技术。 31, 213–219 (2013 年)。
Saunders, C. T. 等人。Strelka:来自测序肿瘤-正常样本对的准确体细胞小变异检出。生物信息学 28, 1811–1817 (2012)。
Deshwar, AG 等人。PhyloWGS:从肿瘤的全基因组测序中重建亚克隆组成和进化。基因组生物学。 16, 35 (2015).
确认
作者感谢患者及其家人参与这项临床试验并捐赠组织用于研究。我们感谢 MSK 斯隆肝胆健康中心、精准病理学生物样本库中心和分子肿瘤学中心的所有临床研究人员。这项工作得到了 Olayan 慈善基金会和 The Tow 基金会的部分支持,并得到了 Stand Up to Cancer Convergence Award(BDG 和 V.P.B.)、NIH U01 CA224175胰腺癌微环境网络癌症登月奖(V.P.B.)、Ben 和 Rose Cole 慈善 PRIA 基金会(V.P.B.)、 Mark 基金会 ASPIRE 奖(B.D.G.)、William H. Goodwin 和 Alice Goodwin 以及联邦癌症研究基金会和 MSK 实验治疗中心(V.P.B. 和 B.D.G.)、MSK 实验免疫肿瘤学中心(PG)和美国-意大利癌症基金会博士后研究奖学金(MM)。综合基因组学核心的服务由 NCI 癌症中心支持补助金 (CCSG, P30 CA08748)、Cycle for Survival 以及 Marie-Josée 和 Henry R. Kravis 分子肿瘤学中心资助。MSK 的团队赞助了这项临床试验。该项目得到了 imCORE 网络、基因泰克、BioNTech、Stand Up To Cancer、Lustgarten 基金会和美国国家癌症研究所胰腺癌微环境网络的支持。
道德宣言
利益争夺
L.A.R.、Z.M.S.、B.D.G. 和 V.P.B. 是与抗原交叉反应性和追踪疫苗诱导的 T 细胞克隆相关的专利申请的发明人。B.D.G. 和 V.P.B. 是新抗原质量建模专利申请的发明人。L.A.R. 是与溶瘤病毒疗法相关的专利的发明者。B.D.G. 曾因默克、百时美施贵宝和中外制药的演讲活动而获得酬金;已获得 Bristol Meyers Squibb、Merck 和 ROME Therapeutics 的研究资助;并曾担任 Darwin Health、Merck、PMV Pharma、Shennon Biotechnologies、Synteny 和 Rome Therapeutics 的薪酬顾问,他是 Rome Therapeutics 的联合创始人。VPB 报告了 Genentech 的酬金和研究支持以及 Bristol-Myers Squibb 的研究支持。A.S.E 从 Up-To-Date 获得版税。A.V. 报告了来自 Lilly、Verastem、BioMed Valley Discoveries、Bristol-Myers Squibb 和 Silenseed 的研究资金。A.C.W. 报告以下内容:Histosonics,咨询和 Ipsen,临床试验资金。EMO 向该机构报告的研究资金来自:基因泰克/罗氏、BioNTech、阿斯利康、Arcus、Elicio、帕克研究所、NIH/NCI、消化保健和突破癌症;通过数据和安全监测委员会 (DSMB) 咨询:Arcus、Alligator、Agenus、BioNTech、Ipsen、Merck、Moma Therapeutics、Novartis、Syros、Leap Therapeutics、Astellas、BMS、Fibrogen、Revolution Medicine、Merus Agios(配偶)、Genentech-Roche(配偶)、Eisai(配偶)和 Servier(配偶)。J.D. 拥有 Alnylam Pharmaceuticals、Arrowroot Acquisition 和 Ionis Pharmaceuticals 的股票。T.M. 是 IMVAQ Therapeutics 的联合创始人并持有股权;是 Immunos Therapeutics、ImmunoGenesis 和 Pfizer 的顾问;获得 Bristol-Myers Squibb、Surface Oncology、Kyn Therapeutics、Infinity Pharmaceuticals、Peregrine Pharmaceuticals、Adaptive Biotechnologies、Leap Therapeutics 和 Aprea 的研究支持;并拥有与溶瘤病毒治疗、基于甲细胞病毒的疫苗、新抗原建模、CD40、GITR、OX40、PD-1 和 CTLA-4 相关的申请专利。JDW 是 Apricity、CellCarta、Ascentage Pharma、阿斯利康、Bicara Therapeutics、勃林格殷格翰、百时美施贵宝、第一三共、Dragonfly、Georgiamune、Imvaq、Larkspur、Psioxus、Recepta、Tizona 和 Sellas 的顾问。JDW 获得 Bristol-Myers Squibb 和 Sephora 的资助和研究支持。JDW 持有 Apricity、Arsenal IO、Ascentage、Imvaq、Linneaus、Georgiamune、Maverick 和 Tizona Therapeutics 的股权。W.P. 报告了来自以下机构的研究资金:默克、安斯泰来、Miracogen 和 Amgen;咨询或顾问委员会活动:安斯泰来和 EXACT Therapeutics;继续医学教育 (CME) 的酬金来自:美国医师研究所和诚信。O.T. 和 US 是 BioNTech 的联合创始人、管理委员会成员和员工。E.D.、L.M. 和 F.M. 是 BioNTech 的员工。I.R.、M.Y. 和 I.M. 是 Genentech 的员工。其他作者声明没有竞争利益。
同行审查
同行评审信息
Nature 感谢 Robert Seder、Neeha Zaidi 和其他匿名审稿人对这项工作的同行评审做出的贡献。
其他信息:
出版商注施普林格·自然 (Springer Nature) 对已发布的地图和机构隶属关系中的管辖权主张保持中立。
扩展数据图表和表格
扩展数据 图 1:在 3 年随访中,Autogene cevumeran 但不相关 atezolizumab 反应或肿瘤大小与 RFS。
a,先前报道的试验示意图1.转载于 CC BY 4.0:https://creativecommons.org/licenses/by/4.0/。b,由离体 IFNγ ELISpot 确定的自体基因头孢乌梅兰(疫苗)免疫反应分层的标志性时间(最后一次疫苗初免剂量)的无复发生存期 (RFS)1 (左)和按抗 PD-L1 (atezolizumab) 反应(中)和中位原发肿瘤大小(右)分层的手术 RFS。HR、风险比和 95% 置信区间。黑色刻度线、审查点。n 是个体患者的数量。P 值通过双尾对数秩检验 (b)。
扩展数据图 2 Autogene cevumeran 在启动但不增强时诱导从头 T 细胞克隆。
a、b,由 CloneTrack 鉴定的疫苗诱导的 T 细胞克隆在引物 (a) 和增强 (b) 处。患者 5 未接受加强剂量,被排除在 (b) 之外。黑色、蓝色、绿色、黄色符号分别表示手术、atezolizumab、疫苗接种、mFOLFIRINOX 时间。水平线,克隆检测阈值;垂直线,重现时间。线条颜色表示调整后的 P 值以进行从头扩展。n 是克隆数。通过修改的双尾 Fisher 精确检验 (a, b) 的 P 值。
扩展数据 图 3 CloneTrack 鉴定的 Autogene 头孢乌梅兰诱导的克隆包含新抗原特异性克隆。
a-d,为了鉴定新抗原特异性 T 细胞克隆,我们在疫苗接种后的不同时间点用重叠的新肽池刺激了 PBMC。然后我们纯化了 CD8CD107a++/−T 细胞和已鉴定的活化 (CD107a) 细胞比例高于灭活 (CD107a) 细胞的克隆作为体外新抗原特异性 T 细胞克隆(NA 特异性)(如前所述+–1; 方法)。对于选定的克隆和患者,我们通过 TCR 克隆 (NA 映射) 验证了新表位特异性。a, 检测示意图。b,c,维恩图显示了单个 (b) 和所有 (c) n 中 CloneTrack 克隆(绿色维恩图)、NA 特异性克隆(鉴定为新抗原特异性的克隆,体外疫苗新抗原特异性 T 细胞激活,如 a,粉红色维恩图)和 NA 定位克隆(通过 TCR 克隆映射到新表位的克隆,如 a,黑色维恩图)= 8 个响应者。轨迹图/维恩图阴影显示了由 CloneTrack 识别并经实验验证的克隆的体内纵向膨胀/收缩(维恩图中的蓝色阴影),由 CloneTrack 识别但未经实验验证(维恩图中的绿色阴影),以及经实验验证但未被 CloneTrack 发现(维恩图中的粉红色阴影)。流式细胞术 (b) 显示 4-1BB 在与 HLA 匹配的、neo 或不相关的肽脉冲抗原呈递细胞一起培养的新抗原特异性 TCR 转导的 CD8 T 细胞(鉴定为 a、NA 定位克隆中)上表达。细胞在活的 CD3CD8 TCR 转导细胞上设门。* 表示 CloneTrack 克隆。(d) 组织中所含的 NA 特异性和 NA 定位克隆(如 c)的百分比。黑色、蓝色、绿色和黄色符号分别表示手术、atezolizumab、疫苗接种和 mFOLFIRINOX 时间。n 是克隆数。+++–
扩展数据图 4 自基因 cevumeran 诱导的 CloneTrack 克隆的动力学、半衰期和寿命。
a,CloneTrack 克隆的自基因 cevumeran 引发剂量数(如扩展数据图 1 所示)。2a) 在血液中达到峰值扩增。红线,达到峰值扩展的中位剂量。b,在 (a) 中,以单个 CloneTrack 克隆在峰值扩增时表示的个体患者 T 细胞库的百分比。c,反应者中每百万个 T 细胞 (n = 8) 中所有 CloneTrack 克隆细胞随时间变化的纵向平均数量。圆圈,意思是;误差线、s.e.m. d、CloneTrack 克隆在引物和增强后出现的峰值扩展(顶部)和半衰期(底部)。e,(上)代表估计的疫苗初免和加强后的轨迹。(下)每个患者每个克隆的估计轨迹的指数拟合的均方根误差 (RMSE)。虚线表示观测到的测量值和估计的测量值之间的平均估计差值相差 2 倍(红色)或 10 倍(黑色)。f,疫苗诱导的初免后(上)和加强后(下)膨胀峰值频率与估计寿命(左)和半衰期(右)的相关性。g,CloneTrack 克隆的半衰期(上)和寿命(下),在初免(左)和增强(右)后具有一个或多个共扩增克隆。h,CloneTrack 克隆半衰期(上)和寿命(下)在单位和多位反应基因中引发后(左)和增强(右)。i,体外按定位/未定位新抗原 (NA) 特异性分层的 CloneTrack 克隆的寿命(如扩展数据图 1 所示)。3a-c)。在 d-h 中,n = 8 名患者在初次后,n = 7 名患者在加强后(患者 5 无加强)。n 是克隆数。Kruskal-Wallis 检验 (b)、双尾 Mann Whitney 检验 (d, g, h) 和双尾 Spearman 相关性 (f) 的 P 值。
扩展数据图 5 共享 TCR Vβ 特异性组中的 T 细胞克隆不与自基因 cevumeran 诱导的 CloneTrack 克隆共扩增。
a、生成概率 (P根) 接种疫苗后 > 2 年(持续性/非持久性)持续存在/不持续存在 CloneTrack 克隆。b,共享 TCR Vβ 特异性组中具有患者内部元克隆的 CloneTrack 克隆的百分比。使用 GLIPH2.0 将元克隆鉴定为具有 TCR 序列的克隆型,其 TCR 序列与疫苗反应性克隆 (即 CloneTrack 克隆) 相似35.c,CloneTrack 元克隆和聚合 CloneTrack 克隆的单个纵向轨迹,用于比较。黑色、蓝色、绿色和黄色符号分别表示手术、atezolizumab、疫苗接种和 mFOLFIRINOX 时间。n 是克隆数。双尾 Mann Whitney 检验 (a) 的 P 值。
扩展数据图 6 atezolizumab 诱导的 T 细胞克隆的动力学。
a,CloneTrack 在所有疫苗反应者中鉴定出 Atezolizumab(抗 PD-L1)诱导的 T 细胞克隆。水平线,克隆检测阈值;红框,autogene cevumeran 响应者。黑色、蓝色、绿色和黄色分别表示手术、atezolizumab、疫苗接种和 mFOLFIRINOX 时间。n 是克隆数。P 值通过修改后的双尾 Fisher 精确检验。
扩展数据图 7 Autogene cevumeran 诱导的 CloneTrack 克隆 T 细胞不会长期获得耗竭特征。
a,按患者(左)、CloneTrack 克隆(中)和 CD8 表达(右)分层的所有血液 T 细胞(所有患者,所有时间)的单细胞 RNA 和 TCR 测序的 UMAP。b-d,通过单细胞 RNA/TCR 测序,CloneTrack 克隆 T 细胞在扩增、收缩和记忆期的选择中枢/效应记忆 (b)、组织保留 (c) 和 T 细胞耗竭 (d) 相关基因的小提琴图,如图 2 所示。3a. n 是细胞或克隆的数量。P 值通过双侧成对 Wilcoxon 秩和检验。
扩展数据 图 8:尽管接种后化疗,Autogene cevumeran 诱导的 CloneTrack 克隆仍保留效应表型。
a,按患者(左)和表型(右)分层的 CloneTrack 克隆 T 细胞(所有患者,所有时间)的单细胞 RNA 和 TCR 测序的 UMAP。b,个体反应者中 CloneTrack 克隆随时间变化的表型组成和转换的 PhenoTrack 图。垂直彩色条形显示相应时间每种表型的细胞比例。黑色、蓝色、绿色和黄色矩形分别表示手术、atezolizumab、疫苗接种和 mFOLFIRINOX 时间。c,单个反应者中 CloneTrack 克隆从扩增到收缩(顶部)和收缩到记忆(底部)阶段的表型转换。箭头,单个克隆;箭头碱基的轴接近度,表型中每个克隆的细胞比例;箭头从表型起点指向目的地;箭头宽度、克隆频率。n 是单元格的数量。
扩展数据 图 10 自基因头孢乌梅拉反应者不会获得针对非疫苗新抗原的免疫活性。
a、b、大量 PBMC 体外再攻击后 CD8 和 CD4 T 细胞的纵向细胞因子产生,非疫苗肿瘤新抗原库描述了反应者的个体 (a, 流式细胞术) 和聚合 (b) 数据。流式细胞术在活体、CD3CD56 上设门+++−CD8 细胞(左图)和活细胞,CD3CD56++−CD4 细胞(右)。+
权利和权限
开放获取本文根据 Creative Commons Attribution-NonCommercial-NoDerivatives 4.0 International License 获得许可,该许可允许以任何媒体或格式进行任何非商业用途、共享、分发和复制,前提是您给予原作者和来源适当的署名,提供指向 Creative Commons 许可的链接,并说明您是否修改了许可材料。根据本许可,您无权共享源自本文或其部分的改编材料。本文中的图像或其他第三方材料包含在文章的知识共享许可中,除非在材料的致谢行中另有说明。如果材料未包含在文章的 Creative Commons 许可中,并且您的预期用途未被法律法规允许或超出允许的用途,您将需要直接从版权所有者处获得许可。要查看此许可证的副本,请访问 http://creativecommons.org/licenses/by-nc-nd/4.0/。
Hits: 60