Influenza vaccine strain selection with an AI-based evolutionary and antigenicity model
自然医学(2025)
摘要
当前的疫苗对快速变异的病毒只能提供有限的保护。例如,美国疾病控制与预防中心的估计显示,2012年至2021年间,流感疫苗对美国门诊疾病的总体有效性平均低于40%。此外,疫苗的临床效果只能通过回顾性方式评估。在此,我们提出一种名为VaxSeer的计算机模拟方法,该方法可在病毒在未来流感季节的相对优势背景下,预测候选疫苗与流行病毒的抗原匹配度。基于10年使用测序和抗原性数据进行的回顾性评估,我们的方法相比年度推荐,始终能筛选出与流行病毒具有更好实证抗原匹配度的毒株。最后,我们预测的抗原匹配度估计值与流感疫苗的有效性以及疾病负担的减少呈现出强相关性,这凸显了该框架在推动疫苗筛选过程中的潜力。
主要内容
世界卫生组织(WHO)的专家小组每年会召开两次会议,为即将到来的流感季节推荐疫苗株。为减轻流感带来的疾病负担,该小组的目标是优化“疫苗效力”,其定义为接种疫苗者与未接种疫苗者相比,流感感染几率的降低程度1。每个流感季节结束后,美国疾病控制与预防中心(CDC)等机构会通过观察性的检测阴性研究来评估流感疫苗效力,这些研究涉及有就医记录的门诊患者2。如果所选的疫苗株与流感季节流行的病毒株分布高度匹配,那么该季节灭活流感疫苗的效力可能达到40%-60%3。然而,尽管在预防和监测方面已有数十年的研究,当前的流感疫苗提供的保护仍然有限。美国流感疫苗效力网络的CDC估计显示,在2012年至2021年的10年间,有5年时间里,不同亚型和年龄组的总体疫苗效力降至40%以下(参考文献4、5、6、7、8、9、10、11、12),同时流感相关住院率有所上升(扩展数据图1)。例如,在2014-2015年冬季流感季,疫苗效力仅为19%6。
许多因素会影响疫苗的有效性,包括人群的免疫史以及疫苗生产中使用的平台和配方13、14、15、16、17、18。然而,疫苗株与流行病毒株的抗原匹配尤为重要。这项工作侧重于改善这种抗原匹配,这是疫苗有效的必要条件11、15、19。
抗原匹配可以通过两种类型的数据进行评估。首先,像全球共享流感数据倡议(GISAID)20这样的监测数据集会收集病毒蛋白的序列及其采集时间,提供目标季节中病毒基因型的分布情况。基于这些数据,我们可以计算出某一病毒株的“优势度”,即其在特定季节出现的频率。其次,“抗原性”数据用于衡量疫苗产生的抗体对特定病毒株的抑制能力。例如,世界卫生组织合作中心(WHO CCs)会进行体外检测,如使用感染后 ferret 抗血清的血凝抑制(HI)试验21,以定量分析候选疫苗对流行病毒的抗原性。这两种数据源都有助于评估抗原匹配。具体而言,我们将疫苗的抗原匹配量化为其针对流行病毒株的抗原性平均值,并以各病毒株的优势度为权重。在本文中,我们将这种抗原匹配的衡量指标称为“覆盖分数”。
由于灭活流感疫苗需要6-9个月的生产周期22,我们需要前瞻性地评估候选疫苗株。然而,疫苗选择时的病毒株分布可能与即将到来的流感季节的病毒株分布不同10、23、24(详见扩展数据图1c)。此外,验证所有候选疫苗针对未来季节可能流行的每种病毒的抗原性成本过高,且可用于研究的病毒标本数量有限。像血凝抑制试验这样的体外检测21只能对有限数量的候选疫苗进行,通常少于10种(参考文献25)。
我们假设,利用基于过往季节上述数据训练的机器学习模型,可以前瞻性地预测覆盖率得分。在本文中,我们提出了VaxSeer,它整合了覆盖率得分两个组成部分的预测因子:一个用于优势性,另一个用于抗原性。由于血凝素(HA)蛋白在病毒感染和免疫反应中起着关键作用,我们仅通过其HA蛋白序列来表征疫苗株和病毒株26,27。我们的优势性预测因子用于估计输入的HA蛋白序列在特定季节出现的概率。由于病毒株之间的竞争,优势性会随时间变化,其变化速率取决于蛋白序列中所有突变的综合效应。传统的流行病学研究将这种变化速率估计为单个氨基酸突变独立作用的总和28,29,30,31,32,33,34,35,36,37。这些方法可能无法充分捕捉更高层次的特性,例如蛋白质稳定性或宿主相互作用,而这些特性取决于整个蛋白质序列中残基之间的复杂相互作用38。在本研究中,我们利用蛋白质语言模型和常微分方程(ODE)来自动捕捉蛋白质序列与其优势性之间的关系。尽管现有的蛋白质语言模型假设适应度景观是静态的39,40,41,42,43,但我们的方法考虑了动态的优势性变化,使其更适合于像流感这样快速进化的病毒。除了优势性预测模型外,我们还开发了抗原性预测模型,该模型以一对蛋白质序列(病毒和疫苗)作为输入,并输出其预测的血凝抑制(HI)试验结果。我们的抗原性预测模型能够对任何疫苗-病毒对的HI试验结果进行计算机模拟预测,减少了对耗时的抗原性实验的需求。
我们利用我们的模型,对两种流感亚型(A/H3N2和A/H1N1)进行了为期10年的回顾性评估。我们考察了覆盖评分与美国、欧洲和加拿大三家独立机构估算的真实世界疫苗有效性之间的相关性,以及美国疾病控制与预防中心(CDC)估算的流感疫苗避免的有症状疾病数量和就医次数。随后,我们研究了VaxSeer前瞻性识别具有高抗原匹配度的疫苗毒株的能力。具体而言,我们根据从观察到的优势毒株和实验性血凝抑制(HI)数据中回顾性计算出的经验覆盖评分,将我们提出的疫苗毒株与世界卫生组织(WHO)的年度推荐进行了对比评估。总体而言,我们的评估凸显了VaxSeer在辅助疫苗选择方面的潜力,它能够从庞大的病毒库中优先筛选出高抗原匹配度的候选毒株,供资源密集型的实验室或临床验证使用。
结果
VaxSeer概述
基于预测覆盖评分选择疫苗株
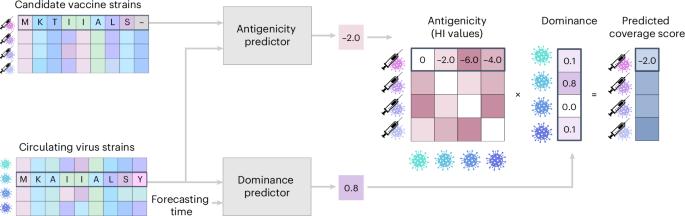
为了在筛选过程中对疫苗毒株进行排名,我们采用计算机模拟方法预测其覆盖评分(图1)。给定一组流行的病毒蛋白和一组候选疫苗蛋白,优势预测器会估算下一个季节每种病毒蛋白的预期优势。此外,抗原性预测器将病毒蛋白与疫苗蛋白的成对序列比对作为输入,预测相应的血凝抑制试验结果。通过将每种候选疫苗针对多种流行病毒的预测抗原性进行平均(以这些病毒的预测优势为权重),这些预测共同提供了该候选疫苗的预测覆盖评分。
训练优势预测器
优势预测器学习血凝素(HA)序列与其优势随时间变化之间的关系,从而能够更准确地预测未来的病毒态势。我们使用疫苗选择时间之前收集的蛋白质序列数据集及其各自的收集日期来训练优势预测器。对于每个蛋白质序列,两个语言模型44会预测初始优势及其变化率,然后将这些预测结果用于常微分方程(ODE)中,以得出该序列在收集时的优势。这两个模型通过使预测的优势与实际的蛋白质分布相匹配来进行训练。
训练抗原性预测器
抗原性预测器通过疫苗和病毒蛋白来学习预测HI试验结果。我们使用疫苗-病毒对的HI试验数据21训练抗原性预测器,所使用的疫苗和病毒蛋白均是在疫苗选择时间之前收集的。HI试验结果与能够抑制特定病毒株的疫苗诱导抗体的量成反比。由于抗原性取决于疫苗株和病毒株,因此抗原性预测器将血凝素(HA)序列对作为输入。为了模拟单个序列内以及序列对之间各位置的关系,抗原性预测器基于用于编码蛋白质多序列比对(MSA)的神经网络架构45。该模型通过将预测的抗原性与实际抗原性进行回归来训练。关于优势预测器和抗原性预测器的更多细节,请参见方法部分。
实验设置
数据构建
我们对VaxSeer在甲型H1N1和甲型H3N2流感亚型上进行了回顾性验证。为了量化每个季节的病毒优势,我们从GISAID下载了带有相关采集时间的流感血凝素(HA)蛋白序列20。在抗原性测量方面,我们根据世界卫生组织合作中心(弗朗西斯·克里克研究所)2003年至2023年2月为流感疫苗成分年度咨询会准备的报告中,收集了基于感染后雪貂抗血清的血凝抑制(HI)试验结果。
为了验证覆盖率得分与疾病结局之间的相关性,我们从美国疾病控制与预防中心(CDC)的年度出版物中收集了历史上使用过的疫苗的流感疫苗有效性数据4、5、6、7、8、9、10、11。这种疫苗有效性是根据美国流感疫苗有效性网络中经医疗护理的门诊急性呼吸道疾病(ARI)患者估算得出的。我们还收集了CDC估算的美国每年因流感疫苗而避免的流感病例数和就医次数46、47。在对疫苗有效性进行分析时,我们排除了没有亚型特异性有效性的疫苗株。我们还排除了2020年和2021年的数据,以避免新冠病毒疫苗可能带来的偏倚48、49,最终得到10种过去推荐的疫苗,这些疫苗有两种亚型的有效性数据。为了进行额外对比,我们还纳入了来自另外两个来源的疫苗有效性估算数据:欧洲的流感监测疫苗有效性项目(I-MOVE)50、51、52、53、54、55以及加拿大的哨兵医生监测网络(SPSN)56、57、58、59、60、61、62、63。I-MOVE根据欧洲多个中心就诊于全科医生的急性呼吸道疾病(ARI)或流感样疾病(ILI)患者来估算流感疫苗有效性。SPSN则根据加拿大社区哨兵医生接诊的流感样疾病(ILI)患者来估算疫苗有效性。在“结果”部分中,除非明确说明(例如SPSN或I-MOVE),否则疫苗有效性均指CDC的估算值。有关有效性估算的更多详细信息,请参见方法和扩展数据表格1。
评估指标
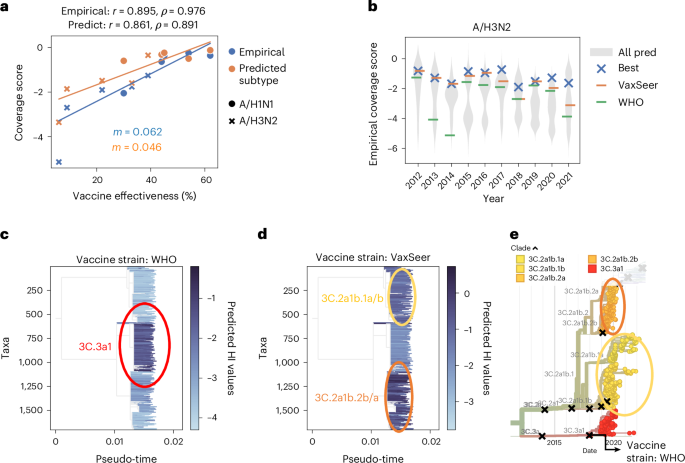
评估不同疫苗选择的主要挑战在于缺乏关于其真实世界有效性的数据。毕竟,我们只能对实际接种给人群的疫苗,衡量其有效性及其对临床终点的影响。相反,我们建议使用一种替代指标,即“经验性”覆盖评分,来反事实评估过去季节中未被选中的疫苗株。经验性覆盖评分是对真实覆盖评分的经验近似,它是利用监测数据(GISAID)中观察到的病毒株频率以及世界卫生组织合作中心的实验性血凝抑制试验结果进行回顾性计算得出的。尽管依赖回顾性数据,但对于在我们的评估中已通过血凝抑制试验充分测试的任何候选疫苗株,都可以计算出经验性覆盖评分。此外,尽管抗原匹配只是众多因素之一,但对于有美国疾病控制与预防中心(CDC)数据的毒株,经验性覆盖评分与疫苗有效性高度相关,皮尔逊相关系数为0.895,斯皮尔曼等级相关系数为0.976,且两者的P值均<0.001(图2a,蓝色)。在独立的欧洲和加拿大有效性研究中,也观察到经验性覆盖评分与疫苗有效性之间存在正相关(扩展数据图2)。根据美国疾病控制与预防中心的估计,经验性覆盖评分还与疫苗在美国预防的疾病负担(有症状疾病和就医次数)呈正相关(扩展数据图3a、b)。疫苗有效性、经验性覆盖评分和预测覆盖评分的定义在扩展数据表格2中进行了区分。
a,经验性和预测性覆盖分数均与美国疾病控制与预防中心(CDC)估计的真实世界疫苗有效性呈正相关(经验性覆盖分数的双侧斯皮尔曼等级相关系数ρ,P=1.46×10⁻⁶;预测性覆盖分数的双侧斯皮尔曼等级相关系数ρ,P=0.0005;经验性覆盖分数的双侧皮尔逊相关系数r,P=0.0005;预测性覆盖分数的双侧皮尔逊相关系数r,P=0.0014;以及线性回归斜率m)。由于经验性覆盖分数与疫苗有效性有很强的相关性,因此它可作为评估疫苗选择的替代指标。b,对于A/H3N2亚型,VaxSeer选择的疫苗株比世界卫生组织(WHO)推荐的疫苗株具有更高的经验性覆盖分数。单侧威尔科克森符号秩检验证实了这种改进的统计学显著性(P=4.1×10⁻⁵)。VaxSeer经常选择经验性覆盖分数最高的毒株。小提琴图展示了所有候选疫苗(包括那些实验性覆盖度低的疫苗)的预测性覆盖分数分布。c、d,2019年北半球流感季流行的A/H3N2亚型毒株相对于WHO推荐疫苗(c)和VaxSeer推荐疫苗(d)的预测抗原性(血凝抑制值,HI值)。我们推荐的疫苗比WHO推荐的疫苗覆盖更多种类的流行病毒(3C.2a1b.1a/b和3C.2a1b.2b/a),而WHO推荐的疫苗仅覆盖3C.3a1。红色圆圈包含3C.3a1分支的病毒,黄色和橙色圆圈分别代表3C.2a1b.1a/b分支和3C.2a1b.2b/a分支的病毒。e,2019年北半球流感季期间及之后A/H3N2亚型的系统发育树,由nextflu78(2024年11月21日版本)构建。每个点代表一个病毒株,黑色十字表示WHO选择的疫苗株。2019年冬季流感季选择的疫苗株用箭头标记。VaxSeer推荐的疫苗株覆盖了优势分支(3C.2a1b.2a/b和3C.2a1b.1a/b)。此外,如右上角灰色分支所示,3C.2a1b.2a分支在后续季节中持续扩散。All pred,所有候选疫苗株的预测性覆盖分数。
回顾性评估设置
我们的评估涵盖2012–2021年的冬季,每年从10月开始,持续到次年3月。由于疫苗有效性数据的可获得性,我们仅在冬季进行评估。根据世界卫生组织(WHO)的推荐时间表,我们使用在季节开始前8个月(2月1日之前)收集的毒株和进行的血凝抑制(HI)试验来训练模型。为了构建候选疫苗毒株和流行病毒毒株集合,我们考虑了过去3年内至少分离出5次的所有病毒毒株,这与WHO推荐的疫苗策略相似。例如,为了评估2021–2022年冬季(2021年10月至2022年3月),我们使用2021年2月1日之前收集的所有数据训练模型,并预测2018年2月至2021年2月期间分离出的候选疫苗毒株的覆盖率得分。为确保对这些候选疫苗毒株的实证覆盖率得分进行准确评估,我们仅评估在目标季节中,针对至少40%的流行病毒序列有HI试验结果的疫苗。共有51种A/H3N2候选疫苗和50种A/H1N1候选疫苗参与了比较。预测的覆盖率得分、实证覆盖率得分以及用于计算实证覆盖率得分的病毒样本百分比均列于补充表1中。关于数据构建和评估设置的更多细节,请参见方法部分。
基于预测覆盖评分的疫苗选择
如图2b和扩展数据图3c所示,根据我们模型的预测覆盖分数选择的疫苗株,在使用经验覆盖分数评估时,在A/H1N1的10年中有6年、在A/H3N2的10年中有9年表现优于世界卫生组织(WHO)的推荐。在其余大多数年份中,我们的推荐与WHO的推荐相似。总体而言(两种亚型,10年),我们的预测在经验覆盖分数上比WHO的推荐有统计学上的显著改善(单侧Wilcoxon符号秩检验,P = 4.1×10⁻⁵)。事实上,VaxSeer在A/H1N1的10年中有7年、在A/H3N2的10年中有5年成功推荐了“最佳”疫苗株(经验覆盖分数最高),而WHO的推荐在A/H1N1的10年中仅3年与最佳疫苗株匹配,在A/H3N2的10年中则没有一年匹配。由于WHO合作中心的实验限制,只有一部分候选疫苗经过了广泛测试(超过40%的流感病毒),从而能够评估它们的经验覆盖分数。因此,图2b和扩展数据图3c中选择的毒株仅反映了这些有限的候选疫苗选择。如果我们考虑过去3年中所有流行候选毒株的预测覆盖分数分布(图2b和扩展数据图3c中的小提琴图),会发现有些毒株的分数甚至更高,但未经过WHO合作中心充分的实验验证。这表明可能存在更有效的疫苗株有待发现。
在某些情况下,我们的模型能够比世界卫生组织(WHO)提前一年预测出合适的疫苗毒株。例如,在2016年A/H1N1流感的冬季流行季,尽管WHO推荐的毒株(A/加利福尼亚/07/2009)具有不错的实际覆盖评分,但VaxSeer提出了另一种评分更高的替代毒株(A/密歇根/45/2015,最早于2015年9月7日采集)。值得关注的是,A/密歇根/45/2015在接下来的冬季流行季被WHO纳入了推荐清单。
为了直观展示不同疫苗株的覆盖范围,我们绘制了2019年冬季流行的A/H3N2毒株的预测抗原性。世界卫生组织(WHO)推荐的毒株与一个新兴分支3C.3a1相匹配(图2c)。相比之下,VaxSeer推荐的毒株具有互补的抗原特性,主要覆盖3C.2a1b.1a/b和3C.2a1b.2b/a分支(图2d)。尽管WHO选择的疫苗覆盖了新分支54,但我们的模型倾向于选择对大多数流行分支以及正在进一步扩张的分支(3C.2a1b.2a)有效的疫苗株,如系统发育树所示(图2e)。
预测的覆盖评分与疫苗效力及临床终点的相关性
与疫苗效力的相关性
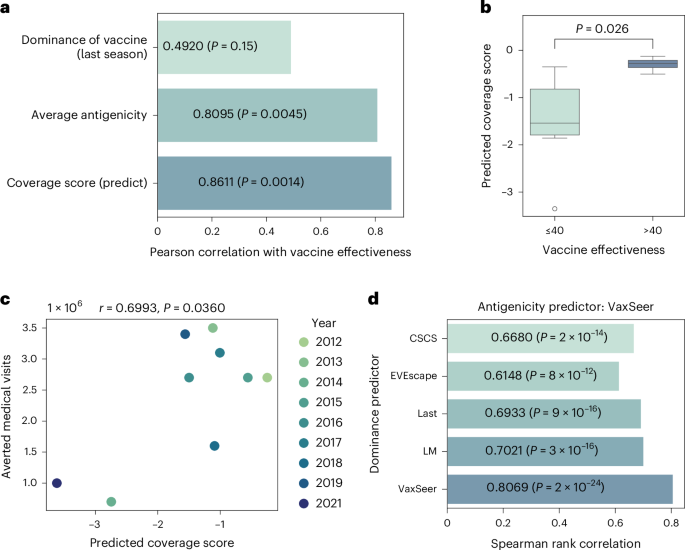
如图2a(橙色)所示,预测的覆盖分数与美国疾病控制与预防中心(CDC)估计的疫苗对门诊急性呼吸道感染(ARI)的有效性显著相关(r = 0.861,P = 0.0014,ρ = 0.891,P = 0.0005)。扩展数据图2显示,在欧洲I-MOVE和加拿大SPSN的疫苗有效性估计中,也一致观察到这种相关性。图3a和扩展数据图4a的进一步分析表明,与仅基于病毒优势或抗原性的替代指标相比,我们结合抗原性和优势以及准确的未来优势预测的方法性能更优。
a,2012至2019年,世卫组织为甲型H1N1和甲型H3N2选择的疫苗株,其疫苗效力与各种评分策略之间的双侧皮尔逊相关系数及相关P值。评估基于n=10个数据点,每个数据点代表世卫组织为特定年份和亚型选择的一种疫苗株,以及来自美国疾病控制与预防中心的相应疫苗效力估计值。我们预测的覆盖率得分显示出最强的相关性。b,效力较高(>40)的疫苗比效力较低(≤40)的疫苗具有更高的预测覆盖率得分。箱线图中的中线表示覆盖率得分的中位数。箱体范围从第一四分位数到第三四分位数,须线覆盖1.5倍四分位距。须线之外的离群值用单个标记表示。图中显示了通过单侧独立t检验计算的P值。c,美国的预测覆盖率得分与通过接种疫苗避免的流感就诊估计次数之间的相关性。文中展示了双侧皮尔逊相关系数(r)及相应的P值(P)。d,基于不同优势预测因子39、42的经验覆盖率得分与预测覆盖率得分之间的双侧斯皮尔曼等级相关系数及相关P值,评估对象为2012至2021年的甲型H1N1(50株)和甲型H3N2(51株)候选疫苗株。每个数据点代表特定年份和亚型的一种独特毒株。鉴于我们的抗原性预测因子,我们的优势模型性能最佳。predict:预测的。
首先,在不考虑抗原性的情况下,上一季疫苗株的优势(图3a中的“上一季疫苗优势”)与疫苗有效性的相关性较弱(皮尔逊相关系数r=0.4920,P=0.15),详见扩展数据图4b。其次,仅使用抗原性而不考虑病毒未来的优势也会产生次优结果。基准的“平均抗原性”方法仅计算一组专家选定病毒的平均血凝抑制(HI)值,其相关性较差(扩展数据图4c)。相比之下,我们预测的覆盖评分与疫苗有效性的相关性最高,这强调了同时对抗原性和未来优势进行建模的必要性。
此外,通过其预测的覆盖率得分对疫苗进行排名,使我们能够区分过去有效性较高(≥40)和较低(<40)的疫苗(图3b)。经单侧独立t检验证实(P = 0.026),有效性较高的疫苗表现出显著更高的预测覆盖率得分。在这里,我们将40%设定为疫苗有效性的阈值,因为当流感疫苗株与流行的流感病毒在抗原上相似时,这一数值通常被认为是有效性的下限3。
与疾病负担减轻的相关性
除了有效性之外,我们进一步分析了预测的覆盖率得分与疾病负担减轻之间的关联,以展示我们方法的潜在影响。美国疾病控制与预防中心(CDC)通过美国接种疫苗避免的就医次数(图3c)和流感病例数(扩展数据图3d)来估算疾病负担的减轻情况⁴⁶,⁶⁴。由于疾病负担减轻的数据并未区分不同亚型,我们结合了两种亚型的预测覆盖率得分,并按每种亚型的感染比例进行加权。2020年因缺乏可用信息被排除在外。如图3c和扩展数据图3d所示,我们预测的覆盖率得分与避免的有症状病例数和就医次数呈正相关(皮尔逊相关系数分别为0.6858和0.6993,P<0.05)。
优势预测器的性能
接下来,我们单独评估我们的优势预测模型。为了优化疫苗的经验覆盖率得分,优势预测模型的性能评估基于其预测经验覆盖率得分的准确性,并结合一个固定的抗原性模型。为了探究VaxSeer中优势建模变化的影响,我们将其与以下基于相同数据训练的静态病毒优势建模基线进行比较。我们首先与一个基于前一季节计算的经验频率来定义优势的基线(“Last”)进行比较。该基线假设变异株的分布在不同季节之间不会发生变化。我们进一步与三个最先进的机器学习模型LM⁴⁰、CSCS³⁹和EVEscape⁴²进行比较,这些模型学习蛋白质的静态适应度景观,并在预测病毒适应度和逃逸能力方面显示出潜力³⁹、⁴¹、⁴²(详见方法部分⁶)。相比之下,我们通过一个由语言模型参数化的时间依赖性常微分方程来定义优势的演变⁴⁴。
我们的结果表明,我们提出的优势预测器与经验覆盖分数的相关性最高(图3d),误差最低(扩展数据图5a,b),结合我们的抗原性预测器,凸显了使用时间模型的重要性。我们还通过其识别未来优势序列的能力展示了我们的优势预测器的卓越性能,如扩展数据图5c所示。扩展数据图5d和扩展数据表3也展示了抗原性模型的比较。
讨论
疫苗是抵御传染病的重要防线。然而,在病毒持续进化的情况下,预测未来病毒株的发展态势并大规模评估候选疫苗的抗原性具有挑战性。在此,我们提出了VaxSeer,这是一个计算机模拟框架,它基于预测的覆盖评分对疫苗株进行排序——覆盖评分是对疫苗与未来病毒的抗原匹配度的前瞻性评估。我们在覆盖评分中考虑了病毒未来的优势度,这一优势度由优势度预测器预测。该预测器使用常微分方程(ODE)来表达优势度随时间的变化,其参数由蛋白质语言模型估计,从而能够基于整个蛋白质序列预测动态优势度。由于公共卫生数据的观察性特征,我们通过计算替代指标(经验覆盖评分)而非基于人群试验的实际疫苗效力来验证VaxSeer。不过,经验覆盖评分与现实世界效力之间的强相关性表明,VaxSeer有望帮助筛选出效力更优的疫苗株。我们的模型可通过两种方式为流感疫苗的选择提供助力。首先,它通过专门考虑未来的病毒分布,为现有的抗原测量提供了补充视角,从而可在选择过程中作为额外的信息来源。其次,作为一种经济高效且计算高效的计算机模拟工具,我们的模型能够快速筛选大量病毒株库,以识别最具潜力的候选株。这些候选株随后可被优先选择,通过双向试验或临床试验等传统实验室技术进行进一步的资源密集型验证。
尽管多种因素都会影响疫苗的最终效果,但我们的研究重点是抗原匹配。我们并未探讨与疫苗生产或宿主相关的其他影响疫苗选择的因素,例如剂量18、65、佐剂选择<bb2>16</bb2>、疫苗平台<bb3>17</bb3>、生产过程中的鸡蛋适应<bb4>66</bb4>、接种时机<bb5>67</bb5>或宿主免疫史<bb6>13</bb6>、<bb7>14</bb7>、<bb8>15</bb8>。尽管未考虑这些因素,我们预测的覆盖率得分仍与疫苗效果表现出合理的相关性,这凸显了其在改善疫苗选择的抗原匹配方面的实用性。此外,我们的方法可以与考虑这些额外因素的方法相结合,以提供对疫苗效果更全面的评估。
在评估抗原匹配度时,我们研究中使用的测序数据可能存在抽样偏差,这给准确确定毒株分布带来了挑战。例如,全球范围内序列的地理分布不均衡,且测序策略可能会随时间发生变化(详见方法)。近年来,人们越来越倾向于直接从原始临床标本中进行测序,而非从经鸡蛋传代或细胞传代的病毒中测序(扩展数据图6a),因为病毒在传代过程中可能会发生突变68。为了评估这些偏差对经验覆盖率得分的影响,我们考察了两个因素:地理位置和传代历史。如扩展数据图7所示,来自不同地点和传代情况的病毒样本得出的经验覆盖率得分保持一致,这表明疫苗的抗原谱对病毒序列中的抽样偏差相对不敏感。尽管如此,获取更多无偏的测序数据仍有助于提高我们预测的准确性。
在本研究中,由于数据可获得性,抗原性是通过一个世界卫生组织合作中心(弗朗西斯·克里克研究所)进行的血凝抑制试验来评估的。这些数据可能会因该研究所采用的方案而产生偏差,而且血凝抑制试验本身也存在一些固有的局限性。首先,它并非测量中和作用,而是通过观察血凝反应的抑制情况来检测抗体与血凝素蛋白的结合。因此,将血凝抑制试验应用于近期的H3亚型变异株存在问题,因为这些毒株的血凝活性有所降低69。其次,所用抗体来自感染后雪貂的抗血清70,这可能与人类群体中的免疫反应不一致13、14、15。第三,疫苗和病毒的传代历史可能会影响血凝抑制试验的结果68、71。尽管存在这些局限性,血凝抑制试验在估算抗原性方面仍然有用,因为从该试验中得出的经验性覆盖分数与疫苗效力呈显著相关性。此外,我们的抗原性预测模型能够从有噪声的血凝抑制数据中学习到有意义的抗原特征,其准确的覆盖分数预测结果便证明了这一点。通过纳入来自更精确检测方法的抗原性数据,如多个实验室的标准化血凝抑制试验、先进的中和试验(如微量中和试验72或基于高内涵成像的中和试验(HINT)73)以及能最大限度减少抗原突变的传代策略74,我们的方法的准确性有望得到进一步提高。
在我们的实验中,我们将候选疫苗株限定为前几个季节流行的毒株,因为抗原性预测器是基于有限数量的现有疫苗株训练的。然而,由于我们的抗原性预测器仅依赖蛋白质序列,原则上它可以计算任何疫苗的覆盖分数,包括那些尚未分离出来或在自然界中不存在的疫苗。通过扩大抗原性数据集中疫苗的多样性,VaxSeer能够探索更大的疫苗空间,使其成为虚拟筛选、功能优化和从头疫苗设计的宝贵工具。尽管如此,还需要进一步研究来评估我们的模型对与训练数据中差异显著的疫苗的泛化能力。
由于在该病毒的数据构建方面付出了大量努力,我们展示了我们的方法在流感病毒研究中的可行性。只要有序列和抗原性数据,这种方法就可以应用于其他大流行性病毒。然而,我们模型的神经网络架构需要充足的训练数据才能有效运行,因此在应用于新发或罕见病毒时可能会受到限制。例如,由于公开可用的抗原性数据有限,验证这种方法在SARS-CoV-2上的效果更具挑战性。尽管如此,通过替代架构,用计算方法预测抗原性和优势性的总体理念仍然是可行的。
可以从多个方向进一步改进这项工作。首先,VaxSeer目前的实现仅考虑了流感病毒的HA蛋白,而研究表明,其他蛋白(如神经氨酸酶)也可能影响病毒的适应性和疫苗的抗原性75、76、77。因此,我们预计对病毒基因组更大部分进行建模,将为疫苗选择提供更全面的信息。其次,我们仅评估了VaxSeer在那些有来自世界卫生组织合作中心(WHO CCs)的充足HI试验数据的疫苗上的性能。有些疫苗株的预测覆盖分数更高,但我们缺乏实验资源来验证它们的实际覆盖分数。第三,我们仅基于HA蛋白序列来预测抗原性。整合HA蛋白的翻译后修饰(如糖基化)可能会提高抗原性预测的准确性71。最后,我们当前的方法仅计算观察到的病毒序列的预测覆盖分数,而没有考虑可能出现的新序列。事实上,在特定的流感季节,超过40%的HA序列在前一年是未曾出现过的(扩展数据图6b)。为了考虑这些新出现的序列,我们可以利用我们的优势预测器作为生成模型,并根据预测的未来分布对病毒序列进行采样。这将使我们能够计算当前和潜在病毒株的预测覆盖分数。遗憾的是,我们没有足够的实验数据来验证这些新病毒的抗原性。
总之,本研究展示了机器学习在协助人类发现更有效的疫苗方面的潜力。
方法
数据集描述
优势
优势预测模型的训练语料来自GISAID。我们下载了2023年3月2日之前提交的394,090条HA序列。我们只保留了来自人类宿主且接近全长(最小长度为553个氨基酸)的HA氨基酸序列。在这些序列中,约67.5%包含性别信息,男女分布几乎均等。样本的地理分布如下:34.8%来自北美洲,29.2%来自欧洲,20.0%来自亚洲,7.1%来自大洋洲,4.9%来自南美洲,4.1%来自非洲。样本的传代历史分布如下:44.0%来自原始标本,31.8%来自细胞系或鸡蛋,24.1%来源未知。宿主年龄分布如下:2.7%小于2岁,13.3%为2-8岁,7.8%为9-17岁,20.9%为18-49岁,7.2%为50-64岁,9.7%为65岁及以上,38.4%没有可用的年龄信息。我们使用了来自全球的HA序列,这与世界卫生组织(WHO)为每种亚型推荐疫苗株的全球任务一致。此外,我们纳入了所有传代历史的序列,因为早年收集的很大一部分病毒来自细胞系传代(扩展数据图6a)。
从2003年10月开始,我们将每6个月划分为一个季节。我们对A/H1N1和A/H3N2这两个亚型分别进行研究。对于每个亚型,我们剔除了HA样本数量少于100个的季节。经过预处理后,获得了28546条A/H3N2的非重复HA序列,以及23736条A/H1N1的非重复HA序列。根据采集时间,这些序列被进一步分为训练集和测试集。具体来说,为了预测某一年冬季(例如,2018年10月至2019年4月的测试集)序列的优势性,我们使用该年2月之前(例如,2018年2月之前)采集的序列数据进行训练。
在训练过程中,我们将序列随机分成训练集和验证集,比例为9:1。最佳模型检查点根据验证损失来选择。用于训练优势预测模型的样本数量详见补充表2。
抗原性
HI试验数据来源于2003年至2023年2月为世界卫生组织(WHO)流感疫苗成分年度咨询会准备的已发表报告。HI试验数据包括病毒株和疫苗株的名称,以及导致血凝抑制的抗体稀释度。我们根据株名从GISAID中检索到了血凝素(HA)序列。无法找到HA序列的毒株被排除在数据集之外。26%的A/H3N2病毒株(6541株中的1700株)和20%的A/H1N1病毒株(7068株中的1398株)被排除。11%的A/H3N2疫苗株(169株中的18株)和5%的A/H1N1疫苗株(77株中的4株)被排除。如果一个株名对应多个HA序列,我们会列出所有可能的HA序列。如果一对疫苗-病毒序列有多个HI试验值,则使用它们的几何平均值。经过处理后,我们获得了A/H1N1的70631个不同疫苗-病毒HA蛋白对以及A/H3N2的63299个不同蛋白对的HI试验结果。A/H1N1共包含3068个不同的病毒序列和109个不同的疫苗序列。A/H3N2共包含2731个不同的病毒序列和255个不同的疫苗序列。疫苗和测试流行病毒的HA蛋白序列通过MMseqs2进行比对(参考文献79)。
HI试验用于确定能够抑制病毒与红细胞结合的最高抗体稀释度21。稀释度越高,表明抗体与病毒的结合能力越强。参考先前的研究80,我们通过相对(对数)稀释度来量化抗原相似性h(v, x):
其中,HI(v, x)是含有疫苗v诱导产生的抗体的血清能够抑制病毒株x的最高稀释倍数(以倍数计)。HI(v, v)是针对生产疫苗v所用的参考病毒v的稀释度。h(v, x)值越高,表明疫苗株v与病毒株x之间的抗原相似性越高。
在训练每年的血凝抑制(HI)预测模型时,我们仅使用2月1日之前收集到序列的疫苗-病毒对。这些配对进一步按8:1:1的比例划分为训练集、验证集和测试集。验证集用于选择最佳的模型检查点,而测试集则用于评估血凝抑制(HI)预测误差。
评估
我们在下面的评估任务中提供了有关数据的详细信息。
疫苗有效性。为了研究疫苗有效性与覆盖率得分之间的关系,我们构建了包含2012年至2021年期间既往推荐疫苗的测试集。这些既往疫苗的有效性评估数据来自三个独立的来源:
- (a)
美国疾病控制与预防中心(CDC):在美国,流感疫苗的有效性是通过美国流感疫苗有效性网络进行评估的。该网络招募在症状出现后7天内,因急性呼吸道感染(新发或加重的咳嗽)到门诊机构(包括急诊科)就诊的参与者(年龄至少6个月)4、5、6、7、8、9、10、11。
- (b)
SPSN(加拿大):SPSN根据加拿大社区哨点医生接诊的、年龄至少1岁且在ILI发病后7天内就诊的患者来估算疫苗有效性56、57、58、59、60、61、62、63。ILI定义为急性起病的发热和咳嗽,并伴有至少一种其他症状,包括咽痛、肌痛、关节痛或极度虚弱。
- (c)
I-MOVE(欧洲):I-MOVE网络涵盖欧洲9个国家的研究站点,其根据因ILI或ARI就诊于全科医生或儿科医生、且在症状出现后8天内采集过样本的患者数据来估算疫苗效力50、51、52、53、54。
我们排除了2020年和2021年的数据,以减轻SARS-CoV-2疫苗影响可能带来的潜在偏差48、49。所使用的疫苗有效性数据及参考文献汇总于扩展数据表1中。
- (a)
经验覆盖率得分。经验覆盖率得分用于两项评估任务:评估预测覆盖率得分与经验覆盖率得分之间的关系,以及评估我们所选疫苗与世界卫生组织推荐疫苗的抗原匹配度。我们构建了一个包含具有可用经验覆盖率得分的候选疫苗的测试集。对于每一年,我们纳入的疫苗株需满足其蛋白质序列在过去3年的GISAID数据集中至少出现5次,且血凝抑制试验结果覆盖该年度至少40%的流行病毒。这使得A/H3N2有51株疫苗株,A/H1N1有50株疫苗株。
疾病负担的减轻。为了研究覆盖率得分与疾病负担减轻之间的相关性,我们构建了一个测试集,其中包含2012年至2022年期间使用的既往推荐疫苗,由于数据不可得,2020年的数据未被纳入。这些疫苗接种所避免的就诊次数和流感患病数的数据来自美国疾病控制与预防中心(CDC)。由于该数据未对亚型进行区分,我们将甲型H1N1和甲型H3N2的覆盖率得分合并,最终得到9个数据点。
覆盖分数的定义
令X表示流行病毒的集合,v表示所讨论的疫苗。未来t季中疫苗v的“覆盖评分(CS)”的数学定义为
其中,pt(x)是t季病毒x的优势度(概率),h(v, x)用于衡量疫苗v与病毒x之间的抗原性。该概率在集合X上进行归一化,即∑x∈Xpt(x)=1。pt(x)值越大,表明病毒x的优势度越高;h(v, x)值越大,表明疫苗v对病毒x的效果越好。覆盖率得分最高的候选疫苗即为我们的算法所推荐的疫苗。
优势预测器
我们的优势预测器旨在模拟蛋白质序列的时间分辨分布。给定一个HA蛋白序列和特定时间作为输入,我们的优势预测器会输出该HA蛋白序列在该特定时间出现的概率(优势)。
给定一个氨基酸序列x ∈ Vᴸ,其长度为L(其中V是所有可能的氨基酸集合,通常|V| = 20),受SIR81模型的启发,我们通过以下常微分方程定义病毒蛋白x的出现频率变化(未归一化的优势度,记为nt(x)):
其中R(x)∈R表示序列x的优势度变化率,exp(b(x))∈R描述初始条件。通过求解该常微分方程,我们可以得到时间t处的频率nt(x),如下
为了得到序列x的优势度(概率)pt(x),我们需要在整个蛋白质序列空间上对nt(x)进行归一化。然而,由于氨基酸组合的数量极其庞大,直接计算概率是不切实际的。相反,我们用“自回归”条件概率来表示该概率44。也就是说,我们建议对条件概率进行建模。
其中xi是序列x中的第i个残基。R(xi∣x<i; θ)和b(xi∣x<i; θ)均由基于Transformer的语言模型44进行参数化。完整蛋白质序列x的概率可以表示为每个残基的条件概率的乘积:
在训练过程中,我们对蛋白质序列对及其收集时间进行采样。语言模型的参数θ基于最大似然估计目标进行优化:
架构
我们使用一个12层的GPT-2模型44来参数化R(xi∣x<i, θ),并使用另一个12层的GPT-2模型来参数化b(xi∣x<i, θ)。优势预测器训练了100个轮次,批处理大小为16,学习率为1×10⁻⁵。
优势预测基线的实现细节
标注为“最后”的基线根据上一赛季计算出的经验频率来定义主导地位。我们纳入了2月1日之前6个月期间收集的序列数据。
CSCS39采用从掩码蛋白质语言模型计算出的突变概率和功能差异来评估蛋白质的适应性以及从抗体中逃逸的能力。
EVEscape42对单个突变的评分通过结合三种信息来源得出:用于适应性预测的深度生成模型、关于HA蛋白的结构信息以评估抗体结合潜力(每个残基位置的加权接触数),以及突变残基与野生型残基之间在电荷和疏水性方面的化学距离。我们使用EVEscape42代码库,在与我们相同的数据集上训练模型。H3和H1蛋白的结构来自蛋白质数据库,其中A/H1N1使用1RVX,A/H3N2使用4FNK。
语言模型以静态方式对序列的主导性进行建模。与公式(5)类似,氨基酸序列x的可能性在不使用收集时间信息的情况下进行自回归分解:
其中F(xi∣x<i; θ)是GTP-2输出的logits(参考文献44)。
抗原性预测器
我们的抗原性预测器是使用12层MSA Transformer45构建的,带有一个线性输出层,用于回归公式(1)中定义的HI值。该模型的训练批次大小为32,学习率为1×10⁻⁵,训练步数为150,000步。
在我们的消融研究中,我们既考虑了实验得出的基线,也考虑了简单的机器学习基线。在BLOSUM基线中,对于2月1日之前收集的且有可用实验HI结果的疫苗-病毒对,我们使用它们的实验结果。对于没有可用实验HI结果的疫苗-病毒对,我们会搜索2月1日之前收集的、在数据集中有HI值测试结果的最相似疫苗-病毒对,并将它们的平均值作为估计的HI结果。相似度是根据BLOSUM62矩阵82计算的,两个病毒-疫苗对之间的相似度是病毒序列间相似度与疫苗序列间相似度的总和。
此外,我们还考虑了两个机器学习基线。LR+(参考文献80)是一种线性回归模型,其输入特征包括病毒和疫苗蛋白质序列的氨基酸替换以及它们在每个位置的氨基酸。最后,CNN是一种卷积神经网络,由两个具有64个通道、核大小为15的一维卷积层组成,中间插入两个一维最大池化层。输入是来自疫苗和病毒的HA蛋白序列的两个独热编码表示的拼接。我们使用参考文献83中建议的超参数。
评估指标
我们采用皮尔逊相关系数和/或斯皮尔曼等级相关系数84作为主要指标,评估覆盖率得分与疫苗效力之间、覆盖率得分与疾病负担减轻之间以及预测覆盖率得分与经验覆盖率得分之间的关系。
当通过夏皮罗-威尔克检验(P>0.01)确定两个待检验的数据序列均呈正态分布时,我们采用了皮尔逊相关分析。当数据不呈正态分布时,我们则使用斯皮尔曼等级相关分析。在分析疫苗有效性与覆盖率得分之间的相关性时,我们也报告了斯皮尔曼等级相关结果,以突出它们之间的单调关系。为评估预测覆盖率得分与实际覆盖率得分之间的相关性,我们额外呈现了使用平均绝对误差(MAE)和均方根误差(RMSE)的结果,以便进行更全面的评估。
我们使用经验覆盖率评分来评估不同疫苗株的抗原匹配度。为了评估统计显著性,我们进行了单侧Wilcoxon符号秩检验。我们选择单侧检验而非双侧检验,是因为我们的目标是根据经验覆盖率评分评估VaxSeer相较于以往方法的优势,而非仅仅识别出任何差异。
作为对优势预测器的补充评估,我们比较了不同优势预测方法所预测的前20%序列的出现频率。为了评估我们模型更高频率的统计显著性,我们进行了单侧威尔科克森符号秩检验。
作为对抗原性预测器的补充评估,我们比较了不同抗原性预测方法所预测的血凝抑制(HI)值的平均绝对误差(MAE)。为了评估我们模型误差较低的统计显著性,我们进行了单侧Wilcoxon符号秩检验,并使用Benjamini–Hochberg方法(错误发现率)对多重比较进行校正。
报告摘要
有关研究设计的更多信息,请参见本文所链接的《自然》系列期刊论文报告摘要。
数据可用性
HA序列及其元数据(包括采集时间和毒株名称)来自GISAID(https://gisaid.org/)。本研究中使用的蛋白质的登录号可在我们的GitHub仓库(https://github.com/wxsh1213/vaxseer/blob/main/data/gisaid/ha_acc_ids.csv)中获取。血凝抑制(HI)结果来自弗朗西斯·克里克研究所的全球流感中心实验室,可在其年度报告和中期报告(https://www.crick.ac.uk/research/platforms-and-facilities/worldwide-influenza-centre/annual-and-interim-reports)中查阅。人类流感疫苗成分来自https://gisaid.org/resources/human-influenza-vaccine-composition/。美国的疫苗有效性数据来自美国疾病控制与预防中心(CDC)的研究:https://www.cdc.gov/flu-vaccines-work/php/effectiveness-studies/。通过接种疫苗避免的估计疾病负担可在https://www.cdc.gov/flu/vaccines-work/burden-averted.htm中找到。
代码可用性
用于VaxSeer数据处理、训练和评估的所有模型和代码均可在https://github.com/wxsh1213/vaxseer公开获取。
参考文献
https://www.nature.com/articles/s41591-025-03917-y
Hits: 34